Ejercicio: Manipulación de datos, parte 3: Imputación de valores que faltan mediante el aprendizaje automático
Como ha confirmado player_df.isna().sum() en la unidad anterior, en PER solo se conservan nueve valores que faltan. No se puede usar un promedio simple para imputar valores en esa columna. Pero cierta experiencia al respecto puede ser útil.
El valor PER se calcula a partir de los valores de las nueve columnas que lo preceden en el objeto DataFrame (de GP a REBR). Pero la estadística PER es difícil de calcular (vea los detalles en basketball-reference.com). Por tanto, hará lo que hacen los científicos de datos: crear un modelo para proporcionar una buena aproximación.
Sería recomendable crear un modelo de regresión lineal simple para calcular los valores PER que faltan. Pero como sucede con cualquier estimación, tendrá que preguntarse cuánta precisión ofrece.
Para extraer cierto sentido de la precisión de un modelo, podría usar el aprendizaje automático para dividir los datos en dos subconjuntos: de prueba y de entrenamiento. El subconjunto de entrenamiento es la parte de los datos que se usa para entrenar el modelo. El otro subconjunto se utiliza para probar el modelo. Normalmente, se usa el 75 % de los datos para entrenar el modelo y el 25 % restante para probarlo.
Pero, ¿qué ocurre si la división aleatoria de los datos de prueba y de entrenamiento resulta inusualmente afortunada o desafortunada? Por ejemplo, ¿qué ocurre si ha seleccionado jugadores humanos para entrenar el modelo, pero después ha probado la precisión del modelo en los jugadores del equipo de los Looney? Es probable que esto no genere resultados precisos.
Para evitar este problema, los estadísticos y los científicos de datos usan una técnica denominada validación cruzada. La idea es iterar por el conjunto de datos y dividir los datos de maneras diferentes entre datos de entrenamiento y datos de prueba. El uso repetido de esta técnica debería proporcionarle una idea razonable de cómo funcionará el modelo con nuevos datos, incluso si solo tiene datos limitados con los que trabajar.
En esta imagen se proporciona una visualización del proceso de validación cruzada:
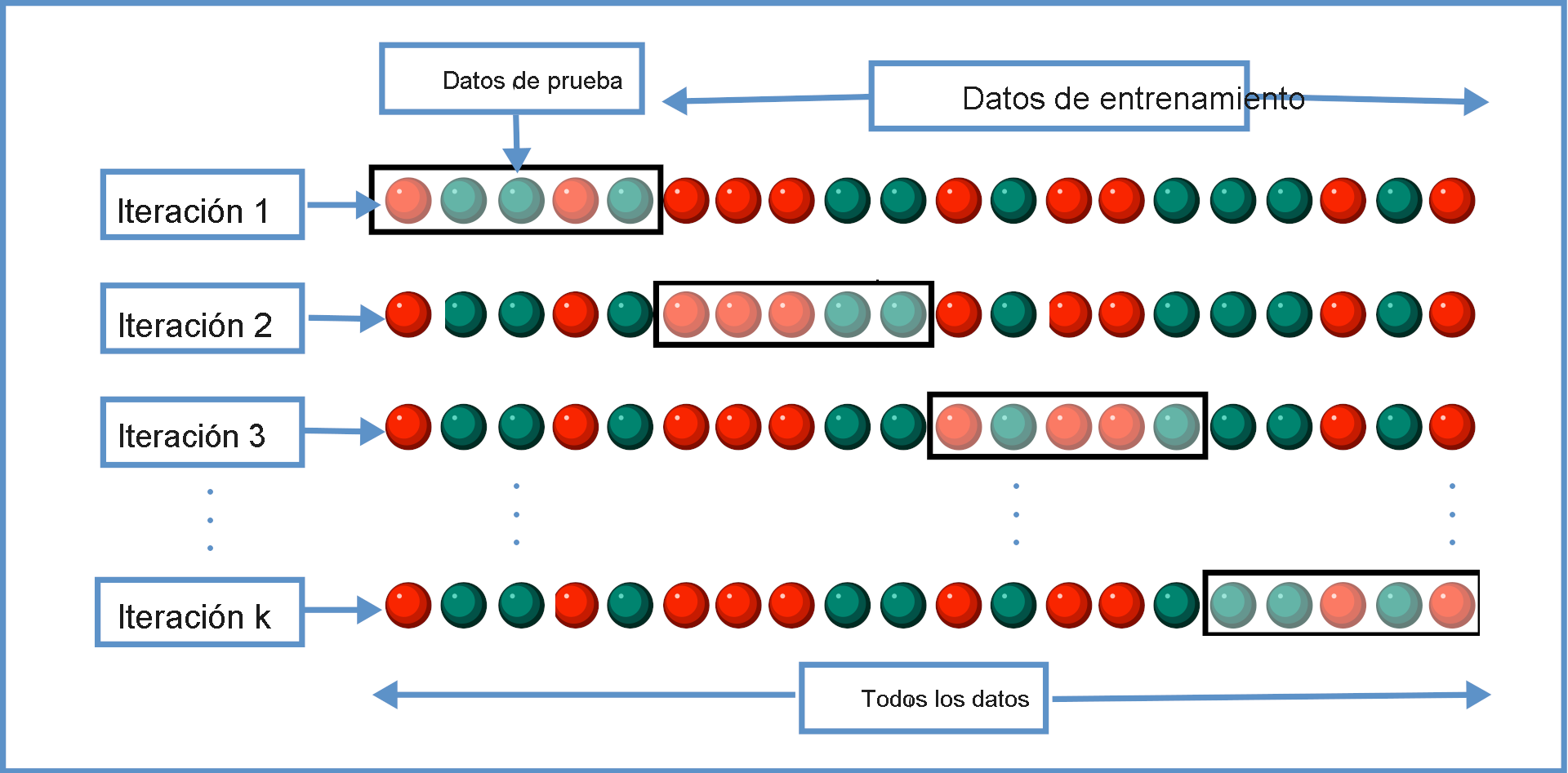
Validación cruzada de las puntuaciones R2 para el modelo
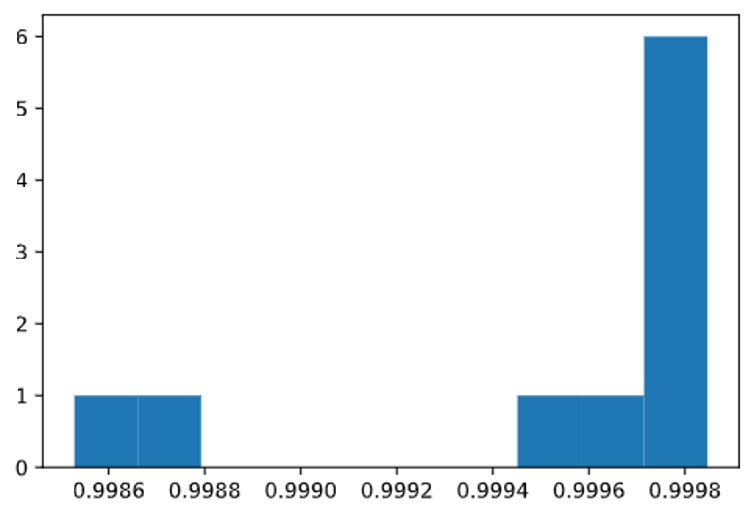
Aquí utilizará una validación cruzada de 10 iteraciones. Es decir, Python iterará 10 veces por los datos, y cada vez se reservará el 10 % de los datos para las pruebas y el entrenamiento se realizará en el 90 % restante. También trazará un histograma de los resultados.
Nota:
Cuando lea el código de Python, tenga en cuenta que, por convención, las predicciones se definen como X. Con el fin de crear el modelo, solo debe usar filas que contengan valores NaN (player_df.dropna(how='any')). Después, use el atributo iloc de DataFrame para seleccionar columnas por número en lugar de por nombre (iloc[:, 4:-1]). Por último, use solo los valores del segmento DataFrame resultante, en lugar del segmento en formato de DataFrame. (Aquí, se prefiere el método to_numpy() al atributo values de DataFrame, de acuerdo a la pandas.DataFrame.values documentación de ).
El código del valor de predicción (y) es similar al código de X, con la excepción de que no tiene que extraer solo los valores.
from sklearn.linear_model import LinearRegression
from sklearn.metrics import r2_score
from sklearn.model_selection import cross_val_score
# Define the variables for the regression model as those rows that have no missing values.
X = player_df.dropna(how='any').iloc[:, 5:-1].to_numpy()
y = player_df.dropna(how='any').iloc[:, -1]
# Define the regression model.
lin_reg = LinearRegression()
# Use the scikit-learn cross-validation function to fit this model 10 times and return the R2 scores.
scores = cross_val_score(lin_reg, X, y, cv=10, scoring='r2')
# Define the histogram of the scores and copy out information from the histogram.
entries, bin_edges, patches = plt.hist(scores, bins=10);
# Print out the mean and the results from the histogram.
print('Mean r2 score: {:.4f}'.format(scores.mean()))
for i in range(len(entries)):
if entries[i] > 0:
print('{:.0f}% of r2 scores are between {:.4f} and {:.4f}'.format(entries[i]*100/len(entries),
bin_edges[i],
bin_edges[i+1]))
Mean r2 score: 0.9995
10% of r2 scores are between 0.9985 and 0.9987
10% of r2 scores are between 0.9987 and 0.9988
10% of r2 scores are between 0.9995 and 0.9996
10% of r2 scores are between 0.9996 and 0.9997
60% of r2 scores are between 0.9997 and 0.9998
Nota:
Si recibe un error al ejecutar este código, asegúrese de que ha instalado la biblioteca scitkit-learn.
En resumen, el modelo es bueno. La puntuación de R2 es del 99,95 %. La puntuación R2 (es decir, R al cuadrado) indica la cantidad de varianza de datos que captura el modelo. Para los objetivos, esta puntuación representa de forma aproximada la precisión del modelo. Las puntuaciones de R2 más bajas siguen siendo correctas y la mayoría se aproximan al valor máximo de 1. Por tanto, debería sentirse seguro de aplicar este modelo a los valores PER que faltan.
Nota
Las puntuaciones de R2 exactas pueden diferir ligeramente. Cada vez que ejecute este modelo, obtendrá un muestreo aleatorio diferente de los datos por corte entre los diez cortes. Por tanto, es posible que el modelo sea ligeramente mejor o peor entre ejecuciones. Pero los números deberían aproximarse bastante a los del ejemplo.
Ajuste del modelo de regresión para los datos de los jugadores
Ahora ajustará el modelo de regresión a todos los datos. (La regla general consiste en usar la validación cruzada para la selección o evaluación del modelo, pero utilizar todos los datos para crearlo).
# Fit the same regression model, this time using all of the available data.
lin_reg.fit(X, y)
LinearRegression(copy_X=True, fit_intercept=True, n_jobs=None, normalize=False)
Creación de una máscara de filas que usen valores que faltan en el objeto DataFrame
No hay una forma elegante para que fillna() use los resultados del modelo para rellenar los valores PER que faltan. Por tanto, tendrá que usar una máscara.
En pandas, una máscara es un mapa booleano de valores que cumplen una determinada condición o un conjunto de condiciones. Para rellenar solo los valores restantes, la máscara debe tener el siguiente aspecto:
# Create and display a mask of rows in the DataFrame. Rows should contain at least one NaN value.
mask = player_df.isnull().any(axis=1)
mask
0 False
1 False
2 True
3 False
4 False
5 False
6 False
7 True
8 True
9 False
10 False
11 True
12 False
13 False
14 False
15 True
16 False
17 False
18 False
19 False
20 True
21 False
22 False
23 False
24 True
25 False
26 False
27 False
28 False
29 True
30 False
31 False
32 False
33 False
34 False
35 False
36 False
37 True
38 False
39 False
40 False
dtype: bool
Ahora aplique esa máscara solo a las columnas que ha usado como X en el modelo. Use el método loc de DataFrame:
# Apply the mask defined earlier to show the contents of specific columns of rows that contain NaN values.
player_df.loc[mask].iloc[:, 5:-1]
Salida
| GP | MPG | TS% | AST | TO | USG | ORR | DRR | REBR | |
|---|---|---|---|---|---|---|---|---|---|
| 2 | 55,000000 | 36,300000 | 0,605 | 25,7 | 13.9 | 28,1 | 4.5. | 4,9 | 1.8 |
| 7 | 57,000000 | 35,272973 | 0,574 | 24,4 | 11.3 | 26,3 | 5.5 | 5.8 | -2,2 |
| 8 | 61,000000 | 35,600000 | 0,547 | 22,9 | 12,2 | 22,7 | 5.8 | 6.4 | -2,9 |
| 11 | 59,972222 | 31,700000 | 0,584 | 32,4 | 14.7 | 16,5 | 3.2 | 19,0 | 4,1 |
| 15 | 59,972222 | 34,900000 | 0,603 | 26,2 | 11,1 | 36,7 | 3.0 | 14.3 | 5.3 |
| 20 | 61,000000 | 35,272973 | 0,645 | 20,6 | 13.1 | 31.5 | 4.0 | 12.0 | 5.2 |
| 24 | 48,000000 | 35,100000 | 0,569 | 19,0 | 10.9 | 29,2 | 4,6 | 17,7 | 5.2 |
| 29 | 64,000000 | 36,300000 | 0,619 | 30,9 | 15,6 | 34,5 | 5.9 | 18,9 | 14.8 |
| 37 | 64,000000 | 36,500000 | 0,618 | 31.3 | 14,0 | 34,9 | 5.9 | 21,3 | 14.5 |
Ahora ha identificado las nueve filas que tienen un valor NaN en la columna PER y tiene las columnas que se pueden usar en el modelo de Machine Learning para predecir el valor PER.
Uso de la máscara y la máscara ajustada para imputar los valores que faltan finales en el objeto DataFrame
En este caso una máscara es útil porque permite utilizar una vista del DataFrame player_df en lugar de un segmento. Al definir X, ha usado player_df.dropna(how='any').iloc[:, 5:-1].to_numpy(). De forma predeterminada, cuando se llama al método dropna() crea un objeto DataFrame. (Esta característica es el motivo por el que ha usado el parámetro inplace del método en el objeto DataFrame original). Por tanto, los cambios que pueda realizar en el objeto DataFrame X no cambiarán los valores del objeto DataFrame player_df.
La máscara es diferente. Simplemente permite trabajar con un subconjunto del objeto DataFrame player_df. Por tanto, los cambios que realice en el objeto DataFrame mientras aplica la máscara también se aplicarán al objeto DataFrame player_df como un todo.
# Impute the missing values in 'PER' by using the regression model and mask.
player_df.loc[mask, 'PER'] = lin_reg.predict(player_df.loc[mask].iloc[:, 5:-1])
# Recheck the DataFrame for rows that have missing values.
player_df.isna().sum()
ID 0
player 0
points 0
possessions 0
team_pace 0
GP 0
MPG 0
TS% 0
AST 0
TO 0
USG 0
ORR 0
DRR 0
REBR 0
PER 0
dtype: int64
A continuación, revise el objeto DataFrame completo para comprobar si faltan valores.
# Display the entire DataFrame.
player_df
Salida
| ID | player | puntos | possessions | team_pace | GP | MPG | TS% | AST | TO | USG | ORR | DRR | REBR | PER | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | player1 | 1893,0 | 1251,8 | 97,8 | 63,000000 | 33,900000 | 0,569 | 17.2 | 11.5 | 26,1 | 4,7 | 23,3 | 7.8 | 10,900000 |
| 1 | 2 | player2 | 1386,0 | 1282,5 | 110,5 | 58,000000 | 32,500000 | 0,511 | 24,8 | 9.7 | 26,9 | 6.1 | 0.9 | 10,7 | 27,300000 |
| 2 | 3 | player3 | 1405,0 | 1252,3 | 105,8 | 55,000000 | 36,300000 | 0,605 | 25,7 | 13.9 | 28,1 | 4.5. | 4,9 | 1.8 | 16,585893 |
| 3 | 4 | player4 | 1282,0 | 1235,9 | 100,7 | 54,000000 | 37,600000 | 0,636 | 29,5 | 11,0 | 22,3 | 4.8 | 4,6 | 5.6 | 22,350000 |
| 4 | 5 | player5 | 1721,0 | 1254,0 | 105,7 | 59,000000 | 30,500000 | 0,589 | 22,8 | 9.9 | 24,6 | 1.2 | 8,4 | 12.1 | 28,380000 |
| 5 | 6 | player6 | 1004,0 | 1322,4 | 102,1 | 57,000000 | 36,300000 | 0,574 | 20,3 | 13.5 | 31,0 | 1.2 | 20,5 | 3.4 | 9,830000 |
| 6 | 7 | player7 | 1920,0 | 1207,6 | 109,8 | 55,000000 | 37,000000 | 0,573 | 26,0 | 13.1 | 30,7 | 0,0 | 10,3 | 2,6 | 8,710000 |
| 7 | 8 | player8 | 1353,0 | 1348,1 | 112,2 | 57,000000 | 35,272973 | 0,574 | 24,4 | 11.3 | 26,3 | 5.5 | 5.8 | -2,2 | 8,708429 |
| 8 | 10 | player10 | 1468,0 | 1400,9 | 93,7 | 61,000000 | 35,600000 | 0,547 | 22,9 | 12,2 | 22,7 | 5.8 | 6.4 | -2,9 | 3,104597 |
| 9 | 11 | player11 | 1856,0 | 1303,8 | 93,4 | 55,000000 | 33,600000 | 0,563 | 28,9 | 14.7 | 26,7 | 1.4 | 17.3 | 4.3 | 9,670000 |
| 10 | 12 | player12 | 1058,0 | 1231,5 | 99,4 | 63,000000 | 32,800000 | 0,550 | 25,1 | 12.9 | 27,2 | 2.1 | 12.1 | 11.4 | 22,97000 |
| 11 | 13 | player13 | 1452,0 | 1246,1 | 103,6 | 59,972222 | 31,700000 | 0,584 | 32,4 | 14.7 | 16,5 | 3.2 | 19,0 | 4,1 | 3,387587 |
| 12 | 14 | player14 | 1438,0 | 1499,6 | 108,1 | 60,000000 | 35,000000 | 0,569 | 28,6 | 12,2 | 36,2 | 4,9 | 6.0 | 7.4 | 27,570000 |
| 13 | 15 | player15 | 1782,0 | 1329,0 | 104,6 | 51,000000 | 35,272973 | 0,590 | 26,5 | 9.9 | 29,7 | 3.1 | 29,4 | 5.5 | 9,380000 |
| 14 | 16 | player16 | 1276,0 | 1257,5 | 89,7 | 58,000000 | 32,800000 | 0,572 | 15,5 | 16,2 | 23.8 | 3.4 | 15,4 | 6.0 | 9,490000 |
| 15 | 17 | player17 | 1486,0 | 1343,6 | 100,6 | 59,972222 | 34,900000 | 0,603 | 26,2 | 11,1 | 36,7 | 3.0 | 14.3 | 5.3 | 22,612442 |
| 16 | 18 | player18 | 1326,0 | 1542,0 | 107,8 | 52,000000 | 31,800000 | 0,583 | 25,3 | 11.7 | 30.6 | 3.3 | 9.9 | 5.3 | 19,780000 |
| 17 | 19 | player19 | 1535,0 | 1336,2 | 104,0 | 56,000000 | 35,400000 | 0,636 | 27,2 | 10,8 | 29,9 | 0,7 | 5.6 | 7.9 | 30,440000 |
| 18 | 21 | player21 | 1276,0 | 1354,9 | 101,2 | 50,000000 | 35,272973 | 0,560 | 21,4 | 12,4 | 28,6 | 2.6 | 9.3 | 10,3 | 24,230000 |
| 19 | 22 | player22 | 1784,0 | 1287,1 | 101,3 | 59,000000 | 33,800000 | 0,601 | 27,2 | 12,4 | 21,8 | 2.9 | 5.2 | 9.9 | 25,150000 |
| 20 | 23 | player23 | 1729,0 | 1205,0 | 103,8 | 61,000000 | 35,272973 | 0,645 | 20,6 | 13.1 | 31.5 | 4.0 | 12.0 | 5.2 | 21,262780 |
| 21 | 24 | player24 | 1639,0 | 1287,4 | 105,2 | 52,000000 | 35,400000 | 0,570 | 32,7 | 15.0 | 26,6 | 1.6 | 5.0 | 9,4 | 25,120000 |
| 22 | 26 | player26 | 1370,0 | 1384,7 | 103,3 | 59,972222 | 34,200000 | 0,571 | 14.8 | 9.9 | 33,1 | 2.4 | 16.3 | 3.7 | 15,040000 |
| 23 | 27 | player27 | 1497,0 | 1410,9 | 98,3 | 57,000000 | 34,100000 | 0,613 | 15,4 | 10.0 | 29,3 | 3.8 | 5.7 | 6.7 | 25,350000 |
| 24 | 28 | player28 | 1313,0 | 1420,5 | 105,0 | 48,000000 | 35,100000 | 0,569 | 19,0 | 10.9 | 29,2 | 4,6 | 17,7 | 5.2 | 12,920786 |
| 25 | 29 | player29 | 1464,0 | 1353,4 | 103,3 | 63,000000 | 34,400000 | 0,595 | 17.9 | 9.2 | 29,3 | 1.0 | 8,7 | 4,1 | 20,490000 |
| 26 | 31 | tune_squad1 | 2049,0 | 1434,0 | 110,0 | 64,000000 | 38,800000 | 0,619 | 31.5 | 14.9 | 35,5 | 8.3 | 17.6 | 12.8 | 28,440000 |
| 27 | 32 | tune_squad2 | 1795,0 | 1481,8 | 112,1 | 62,000000 | 35,400000 | 0,608 | 31,9 | 14.5 | 32,0 | 6.5 | 22.5 | 12.9 | 23,340000 |
| 28 | 33 | tune_squad3 | 1805,0 | 1509,9 | 108,6 | 64,000000 | 35,400000 | 0,622 | 27,9 | 13.9 | 36,0 | 5.9 | 27,7 | 12,2 | 22,410000 |
| 29 | 34 | tune_squad4 | 1743,0 | 1422,4 | 112,9 | 64,000000 | 36,300000 | 0,619 | 30,9 | 15,6 | 34,5 | 5.9 | 18,9 | 14.8 | 29,858714 |
| 30 | 35 | tune_squad5 | 1963,0 | 1539,1 | 117,4 | 59,972222 | 35,272973 | 0,633 | 32,3 | 16,2 | 34,0 | 5.9 | 19,8 | 13.1 | 27,160000 |
| 31 | 36 | tune_squad6 | 2062,0 | 1505,7 | 111,5 | 59,972222 | 37,000000 | 0,620 | 29,8 | 15,6 | 36,2 | 4,9 | 23,9 | 14.7 | 27,860000 |
| 32 | 37 | tune_squad7 | 1845,0 | 1435,7 | 113,1 | 69,000000 | 36,900000 | 0,634 | 33,2 | 14,0 | 36,5 | 4,1 | 21,5 | 16.4 | 34,260000 |
| 33 | 38 | tune_squad8 | 1778,0 | 1526,4 | 109,3 | 66,000000 | 34,900000 | 0,612 | 30.6 | 15,9 | 35,9 | 5.5 | 18,8 | 13,7 | 28,650000 |
| 34 | 39 | tune_squad9 | 1901,0 | 1444,1 | 109,7 | 67,000000 | 36,500000 | 0,609 | 27,2 | 14.8 | 35,5 | 5.0 | 21,8 | 8,9 | 20,120000 |
| 35 | 41 | tune_squad11 | 2030,0 | 1431,0 | 112,3 | 68,000000 | 37,000000 | 0,618 | 32,5 | 15,3 | 34,5 | 5.7 | 15,7 | 13.2 | 30,070000 |
| 36 | 42 | tune_squad12 | 1631,0 | 1465,7 | 110,1 | 66,000000 | 37,500000 | 0,613 | 28,4 | 14.4 | 35,7 | 6.5 | 20,7 | 14,0 | 28,400000 |
| 37 | 43 | tune_squad13 | 1828,0 | 1507,2 | 112,7 | 64,000000 | 36,500000 | 0,618 | 31.3 | 14,0 | 34,9 | 5.9 | 21,3 | 14.5 | 29,102157 |
| 38 | 44 | tune_squad14 | 1821,0 | 1443,7 | 118,8 | 66,000000 | 36,600000 | 0,609 | 27,3 | 13.5 | 35,8 | 7.0 | 23.8 | 11.5 | 22,960000 |
| 39 | 45 | tune_squad15 | 1740,0 | 1443,9 | 114,1 | 68,000000 | 37,100000 | 0,611 | 26,6 | 15.2 | 29,3 | 8.3 | 17,7 | 11,1 | 21,220000 |
| 40 | 46 | tune_squad16 | 1993,0 | 1459,0 | 112,5 | 59,972222 | 36,900000 | 0,627 | 30,4 | 15.0 | 33,7 | 6.3 | 19,3 | 14,1 | 28,760000 |
Por último, guarde este objeto DataFrame en un archivo CSV. Usará este conjunto datos para tomar decisiones antes y durante el partido en función de los jugadores del equipo, los jugadores que participen actualmente en el partido y las estadísticas del partido actual.
player_df.to_csv('player_data_final.csv', index=False)
Nota:
El parámetro index=False garantiza que el índice que se ha agregado al objeto DataFrame al leer inicialmente el archivo CSV por parte de pandas no se escribe en el archivo CSV.
Ahora debería ver un nuevo archivo CSV en la carpeta space-jam-anl.
© 2021 Warner Bros. Ent. All Rights Reserved.