Início Rápido: Executar um fluxo de trabalho através do serviço Microsoft Genomics
Neste início rápido, você carrega dados de entrada em uma conta de armazenamento de Blob do Azure e executa um fluxo de trabalho por meio do serviço Microsoft Genomics usando o cliente Python Genomics. O Microsoft Genomics é um serviço dimensionável e seguro para análise secundária que consegue processar rapidamente um genoma, a partir de leituras não processadas e ao produzir leituras alinhadas e chamadas variantes.
Pré-requisitos
- Uma conta do Azure com uma subscrição ativa. Crie uma conta gratuitamente.
- Python 2.7.12+, com
pipinstalado epythonno caminho do sistema. O cliente Microsoft Genomics não é compatível com Python 3.
Configurar: criar uma conta do Microsoft Genomics no portal do Azure
Para criar uma conta do Microsoft Genomics, navegue até Criar uma conta do Genomics no portal do Azure. Se ainda não tiver uma subscrição do Azure, crie uma antes de criar uma conta do Microsoft Genomics.

Configure a conta do Genomics com as informações seguintes, conforme mostrado na imagem anterior.
| Definição | Valor sugerido | Descrição do campo |
|---|---|---|
| Subscrição | o nome da subscrição | Trata-se da unidade de faturação dos serviços do Azure. Para obter detalhes sobre a sua subscrição, veja Subscrições |
| Grupo de recursos | MyResourceGroup | Os grupos de recursos permitem-lhe agrupar múltiplos recursos do Azure (conta de armazenamento, conta do Genomics, etc.) num único grupo de gestão simples. Para obter mais informações, veja Grupos de Recursos. Para obter os nomes de grupos de recursos válidos, veja Regras de Nomenclatura |
| Nome da conta | MyGenomicsAccount | Escolha um identificador de conta exclusivo. Para obter os nomes válidos, veja Regras de Nomenclatura |
| Location | E.U.A. Oeste 2 | O serviço está disponível nas regiões E.U.A. Oeste 2, Europa Ocidental e Ásia Sudeste |
Você pode selecionar Notificações na barra de menu superior para monitorar o processo de implantação.

Para obter mais informações sobre o Microsoft Genomics, consulte O que é o Microsoft Genomics?
Configurar: instalar o cliente Microsoft Genomics Python
Você precisa instalar o Python e o cliente msgen Python do Microsoft Genomics em seu ambiente local.
Instalar o Python
O cliente Microsoft Genomics Python é compatível com Python 2.7.12 ou uma versão posterior 2.7.xx. 2.7.14 é a versão sugerida. Pode encontrar a transferência aqui.
Importante
Python 3.x não é compatível com Python 2.7.xx. msgen é uma aplicação Python 2.7. Ao executar msgeno , certifique-se de que seu ambiente Python ativo está usando uma versão 2.7.xx do Python. Você pode obter erros ao tentar usar msgen com uma versão 3.x do Python.
Instale o cliente Python do Microsoft Genomics msgen
Use Python pip para instalar o cliente msgenMicrosoft Genomics . As instruções a seguir pressupõem que o Python2.x já está no caminho do sistema. Se você tiver problemas com pip a instalação não sendo reconhecida, você precisa adicionar Python e a subpasta scripts ao caminho do sistema.
pip install --upgrade --no-deps msgen
pip install msgen
Se você não quiser instalar msgen como um binário em todo o sistema e modificar pacotes Python em todo o sistema, use o –-user sinalizador com pip.
Quando você usa a instalação baseada em pacote ou setup.py, todos os pacotes necessários necessários são instalados.
Cliente Python de teste msgen
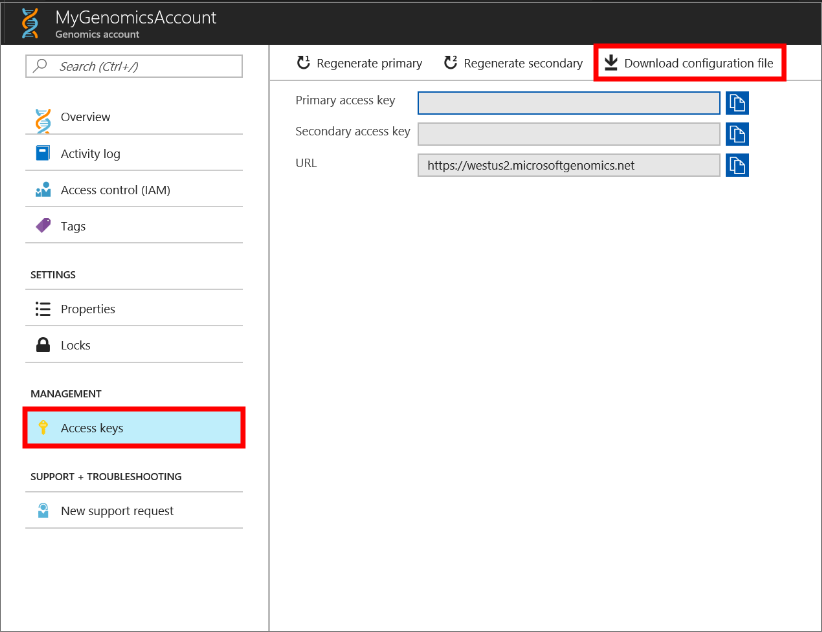
Para testar o cliente Microsoft Genomics, transfira o ficheiro de configuração da sua conta Genomics. No portal do Azure, navegue até sua conta de Genômica selecionando Todos os serviços no canto superior esquerdo e, em seguida, procurando e selecionando Contas de Genômica.

Selecione a conta Genomics que você acabou de criar, navegue até Chaves de acesso e baixe o arquivo de configuração.
Teste se o cliente Microsoft Genomics Python está a funcionar com o seguinte comando
msgen list -f "<full path where you saved the config file>"
Criar uma conta de Armazenamento do Microsoft Azure
O serviço Microsoft Genomics espera que as entradas sejam armazenadas como blobs de blocos numa conta de armazenamento do Azure. Também escreve os ficheiros de saída como blobs de blocos num contentor especificado pelo utilizador numa conta de armazenamento do Azure. As entradas e saídas podem residir em contas de armazenamento diferentes. Se já tiver os dados numa conta de armazenamento do Azure, basta certificar-se de que está na mesma localização da conta do Genomics. Caso contrário, serão incorridas cobranças de saída ao executar o serviço Microsoft Genomics. Se você ainda não tiver uma conta de armazenamento do Azure, precisará criar uma e carregar seus dados. Você pode encontrar mais informações sobre contas de armazenamento do Azure aqui, incluindo o que é uma conta de armazenamento e quais serviços ela fornece. Para criar uma conta de armazenamento do Azure, navegue até Criar conta de armazenamento no portal do Azure.
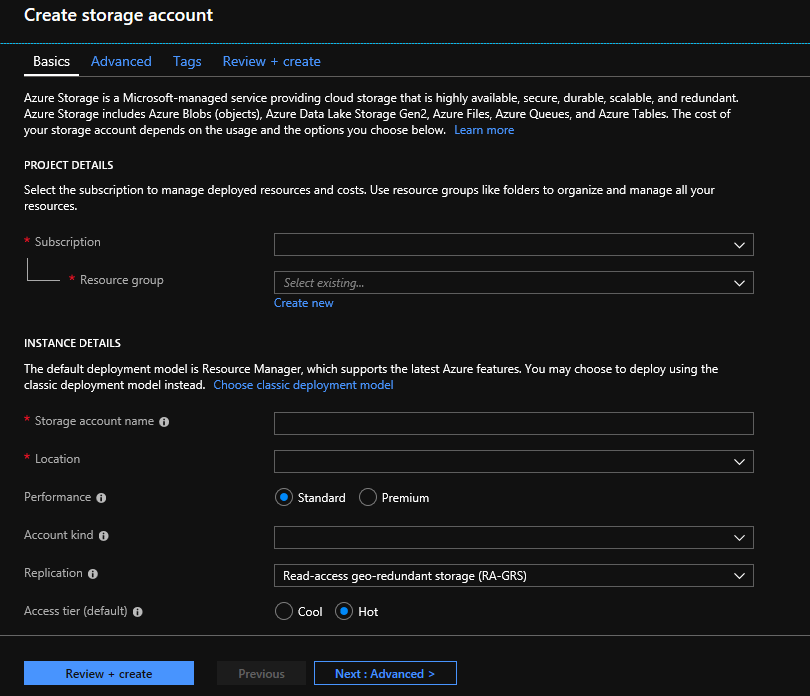
Configure sua conta de armazenamento com as seguintes informações, conforme mostrado na imagem anterior. Use a maioria das opções padrão para uma conta de armazenamento, especificando apenas que a conta é BlobStorage, não de uso geral. O armazenamento de blobs pode ser entre duas e cinco vezes mais rápido para transferências e carregamentos. O modelo de implantação padrão, Azure Resource Manager, é recomendado.
| Definição | Valor sugerido | Descrição do campo |
|---|---|---|
| Subscrição | a subscrição do Azure | Para obter detalhes sobre a sua subscrição, veja Subscrições |
| Grupo de recursos | MyResourceGroup | Pode selecionar o mesmo grupo de recursos que a sua conta de Genómica. Para nomes de grupos de recursos válidos, consulte Regras de nomenclatura |
| Nome da conta de armazenamento | MyStorageAccount | Escolha um identificador de conta exclusivo. Para nomes válidos, consulte Regras de nomenclatura |
| Location | E.U.A. Oeste 2 | Use o mesmo local da sua conta Genomics para reduzir as cobranças de saída e a latência. |
| Desempenho | Standard | A predefinição é Standard. Para obter mais detalhes sobre contas de armazenamento padrão e premium, consulte Introdução ao armazenamento do Microsoft Azure |
| Tipo de conta | BlobStorage | O armazenamento de blobs pode ser entre duas e cinco vezes mais rápido do que para fins gerais para transferências e carregamentos. |
| Replicação | Armazenamento localmente redundante | O armazenamento localmente redundante replica os dados no datacenter na região em que foi criada a conta de armazenamento. Para obter mais informações, veja Replicação do Armazenamento do Azure |
| Camada de acesso | Muito Interessado | Um acesso frequente indica que os objetos na conta de armazenamento serão acedidos com mais frequência. |
Em seguida, selecione Rever + criar para criar a sua conta de armazenamento. Como fez com a criação da sua conta Genomics, você pode selecionar Notificações na barra de menu superior para monitorar o processo de implantação.
Carregar dados de entrada para a conta de armazenamento
O serviço Microsoft Genomics espera leituras finais emparelhadas (arquivos fastq ou bam) como arquivos de entrada. Pode optar por carregar os seus próprios dados ou explorar através dos dados de exemplo disponíveis publicamente fornecidos por si.
Na sua conta de armazenamento, tem de criar um contentor de blobs para os dados de entrada e um segundo contentor de blobs para os dados de saída. Carregue os dados de entrada para o contentor de blobs de entrada. Várias ferramentas podem ser usadas para fazer isso, incluindo o Microsoft Azure Storage Explorer, BlobPorter ou AzCopy.
Executar um fluxo de trabalho através do serviço Microsoft Genomics usando o msgen cliente Python
Para executar um fluxo de trabalho por meio do serviço Microsoft Genomics, edite o arquivo config.txt para especificar o contêiner de armazenamento de entrada e saída para seus dados. Abra o ficheiro config.txt que transferiu da sua conta Genomics. As seções que você precisa especificar são sua chave de assinatura e os seis itens na parte inferior, o nome da conta de armazenamento, a chave e o nome do contêiner para entrada e saída. Você pode encontrar essas informações navegando no portal do Azure para acessar chaves para sua conta de armazenamento ou diretamente do Gerenciador de Armazenamento do Azure.

Se você quiser executar GATK4, defina o process_name parâmetro como gatk4.
Por padrão, o serviço Genomics produz arquivos VCF. Se você quiser uma saída gVCF em vez de uma saída VCF (equivalente a -emitRefConfidence GATK 3.x e emit-ref-confidence GATK 4.x), adicione o emit_ref_confidence parâmetro ao seu config.txt e defina-o como gvcf, como mostrado na figura anterior. Para voltar para a saída VCF, remova-a do arquivo config.txt ou defina o emit_ref_confidence parâmetro como none.
bgzip é uma ferramenta que compacta o arquivo vcf ou gvcf e tabix cria um índice para o arquivo compactado. Por padrão, o serviço Genomics é executado bgzip seguido pela tabix saída ".g.vcf", mas não executa essas ferramentas por padrão para a saída ".vcf". Quando executado, o serviço produz arquivos ".gz" (saída bgzip) e ".tbi" (saída tabix). O argumento é um booleano, que é definido como false por padrão para a saída ".vcf" e como true por padrão para a saída ".g.vcf". Para usar na linha de comando, especifique -bz ou --bgzip-output como true (execute bgzip e tabix) ou false. Para usar esse argumento no arquivo config.txt , adicione bgzip_output: true ou bgzip_output: false ao arquivo.
Envie seu fluxo de trabalho para o serviço Microsoft Genomics usando o msgen cliente Python
Utilize o cliente Python do Microsoft Genomics para submeter o seu fluxo de trabalho com o seguinte comando:
msgen submit -f [full path to your config file] -b1 [name of your first paired end read] -b2 [name of your second paired end read]
Pode ver o estado dos fluxos de trabalho com o seguinte comando:
msgen list -f c:\temp\config.txt
Quando o fluxo de trabalho for concluído, você poderá exibir os arquivos de saída em sua conta de armazenamento do Azure no contêiner de saída que você configurou.
Próximos passos
Neste artigo, você carregou dados de entrada de exemplo no armazenamento do Azure e enviou um fluxo de trabalho para o serviço Microsoft Genomics por meio do msgen cliente Python. Para obter mais informações sobre outros tipos de ficheiros de entrada que podem ser utilizados com o serviço Microsoft Genomics, veja as seguintes páginas: FASTQ emparelhado | BAM | Múltiplos ficheiros FASTQ ou BAM.