Snabbstart: Köra ett arbetsflöde genom Microsoft Genomics-tjänsten
I den här snabbstarten laddar du upp indata till ett Azure Blob Storage-konto och kör ett arbetsflöde via Microsoft Genomics-tjänsten med hjälp av Python Genomics-klienten. Microsoft Genomics är en skalbar, säker tjänst för sekundär analys som snabbt kan bearbeta ett genom, från råläsningar till produktion av anpassade läsningar och variantanrop.
Förutsättningar
- Ett Azure-konto med en aktiv prenumeration. Skapa ett konto utan kostnad.
-
Python 2.7.12+, med
pipinstallerat ochpythoni systemsökvägen. Microsoft Genomics-klienten är inte kompatibel med Python 3.
Konfigurera: Skapa ett Microsoft Genomics-konto på Azure Portal
Om du vill skapa ett Microsoft Genomics-konto går du till Skapa ett Genomics-konto i Azure Portal. Om du ännu inte har en Azure-prenumeration kan du skapa en innan du skapar ett Microsoft Genomics-konto.
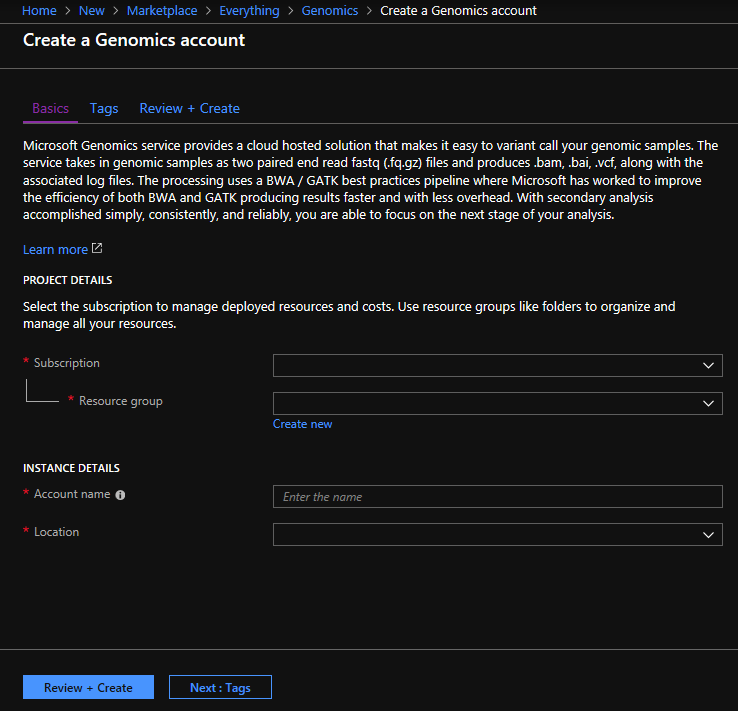
Skapa ditt Genomics-konto med följande information (se föregående bild):
| Inställning | Föreslaget värde | Fältbeskrivning |
|---|---|---|
| Prenumeration | Ditt prenumerationsnamn | Detta är faktureringsenheten för dina Azure-tjänster – mer information om din prenumeration finns under Prenumerationer |
| Resursgrupp | MinResursgrupp | Resursgrupper gör att du kan gruppera flera Azure-resurser (lagringskonto, Genomics-konto, o.s.v.) i en enda grupp för enkel hantering. Mer information finns i Resursgrupper. Information om giltiga resursgruppnamn finns under Namngivningsregler |
| Kontonamn | MittGenomicsKonto | Välj ett unikt konto-ID. Se Namngivningsregler för giltiga namn |
| Location | USA, västra 2 | Tjänsten är tillgänglig i USA, västra 2, Europa, västra och Sydostasien |
Du kan välja Meddelanden på den översta menyraden för att övervaka distributionsprocessen.

Mer information om Microsoft Genomics finns i Vad är Microsoft Genomics?
Konfigurera: Installera Microsoft Genomics Python-klienten
Du måste installera både Python- och Microsoft Genomics Python-klienten msgen i din lokala miljö.
Installera Python
Microsoft Genomics Python-klienten är kompatibel med Python 2.7.12 eller senare version 2.7.xx. 2.7.14 är den föreslagna versionen. Du hittar nedladdningen här.
Viktigt
Python 3.x är inte kompatibelt med Python 2.7.xx.
msgen är ett Python 2.7-program. När du kör msgenkontrollerar du att din aktiva Python-miljö använder en 2.7.xx-version av Python. Du kan få fel när du försöker använda msgen med en 3.x-version av Python.
Installera Microsoft Genomics Python-klienten msgen
Använd Python pip för att installera Microsoft Genomics-klienten msgen. Följande instruktioner förutsätter att Python2.x redan finns i systemsökvägen. Om du har problem med pip att installationen inte känns igen måste du lägga till Python och undermappen skript i systemsökvägen.
pip install --upgrade --no-deps msgen
pip install msgen
Om du inte vill installera msgen som en systemomfattande binärfil och ändra systemomfattande Python-paket använder du –-user flaggan med pip.
När du använder den paketbaserade installationen eller setup.py installeras alla nödvändiga paket.
Testa msgen Python-klienten
Om du vill testa Microsoft Genomics-klienten laddar du ned konfigurationsfilen från ditt Genomics-konto. I Azure Portal navigerar du till ditt Genomics-konto genom att välja Alla tjänster längst upp till vänster och sedan söka efter och välja Genomics-konton.
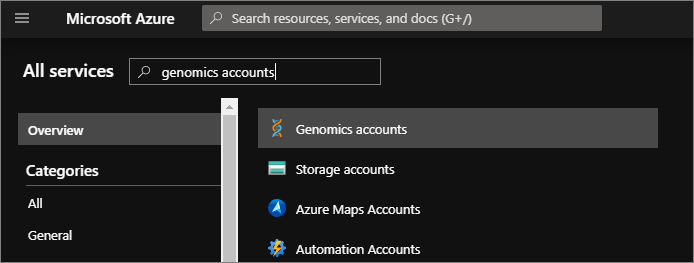
Välj det Genomics-konto som du nyss skapade, navigera till Åtkomstnycklar och ladda ned konfigurationsfilen.
Kontrollera att Microsoft Genomics Python-klienten fungerar med följande kommando
msgen list -f "<full path where you saved the config file>"
Skapa ett Microsoft Azure Storage konto
I Microsoft Genomics-tjänsten förväntas indata lagras som blockblobar i ett Azure Storage-konto. Utdatafilerna skrivs också som blockblobar till en container som angetts av användaren i ett Azure Storage-konto. In- och utdata kan finnas i olika lagringskonton. Om du redan har data i ett Azure Storage-konto behöver du bara se till att det finns på samma plats som Genomics-kontot. I annat fall debiteras utgående avgifter när du kör Microsoft Genomics-tjänsten. Om du ännu inte har ett Azure Storage-konto måste du skapa ett och ladda upp dina data. Du hittar mer information om Azure Storage-konton här, inklusive vad ett lagringskonto är och vilka tjänster det tillhandahåller. Om du vill skapa ett Azure Storage-konto går du till Skapa lagringskonto i Azure Portal.
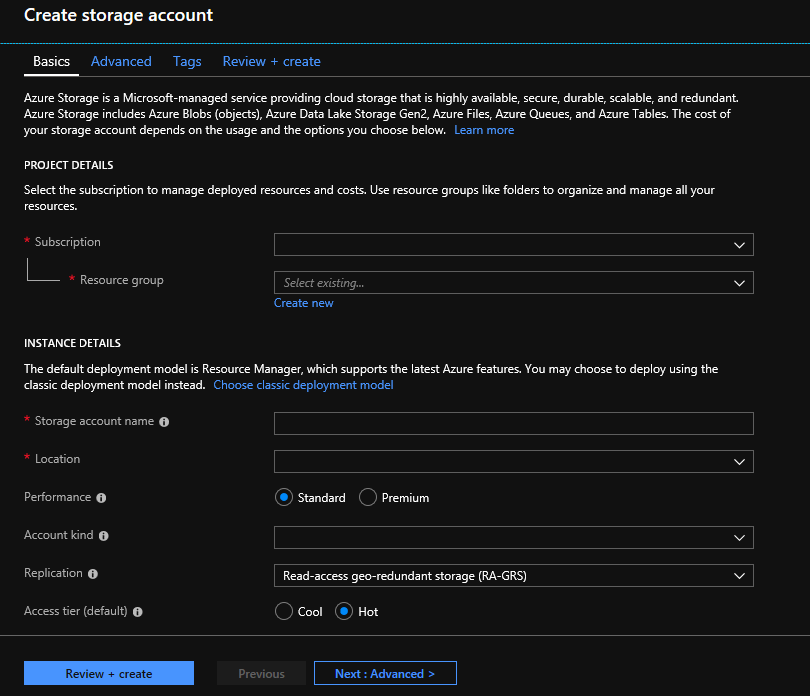
Konfigurera ditt lagringskonto med följande information, enligt föregående bild. Använd de flesta standardalternativen för ett lagringskonto och ange endast att kontot är BlobStorage, inte generell användning. Blob-lagring kan vara 2–5 gånger snabbare för ned- och uppladdningar. Standarddistributionsmodellen, Azure Resource Manager, rekommenderas.
| Inställning | Föreslaget värde | Fältbeskrivning |
|---|---|---|
| Prenumeration | Din Azure-prenumeration | Mer information om din prenumeration finns i Prenumerationer |
| Resursgrupp | MinResursgrupp | Du kan välja samma resursgrupp som ditt Genomics-konto. Giltiga resursgruppsnamn finns i Namngivningsregler |
| Lagringskontonamn | MittLagringskonto | Välj ett unikt konto-ID. Giltiga namn finns i Namngivningsregler |
| Location | USA, västra 2 | Använd samma plats som platsen för ditt Genomics-konto för att minska utgående avgifter och minska svarstiden. |
| Prestanda | Standard | Standardinställningen är Standard. Mer information om standard- och premiumlagringskonton finns i Introduktion till Microsoft Azure Storage |
| Typ av konto | BlobStorage | Blob-lagring kan vara 2–5 gånger snabbare än lagring generell användning för ned- och uppladdningar. |
| Replikering | Lokalt redundant lagring | Med lokalt redundant lagring replikeras dina data i datacentret i den region där du har skapat ditt lagringskonto. Mer information finns i Azure Storage-replikering |
| Åtkomstnivå | Frekvent | Frekvent åtkomst indikerar att objekten på lagringskontot kommer att användas oftare. |
Välj sedan Granska + skapa för att skapa ditt lagringskonto. Precis som när du skapade ditt Genomics-konto kan du välja Meddelanden i det översta menyfältet för att övervaka distributionsprocessen.
Ladda upp indata till ditt lagringskonto
Microsoft Genomics-tjänsten förväntar sig parkopplade slutläsningar (fastq- eller bam-filer) som indatafiler. Du kan välja att antingen ladda upp dina egna data eller utforska med offentligt tillgängliga exempeldata som du får.
I ditt lagringskonto måste du skapa en blob-container för dina indata och en andra blob-container för dina utdata. Ladda upp indata till blob-containern för indata. Olika verktyg kan användas för att göra detta, till exempel Microsoft Azure Storage Explorer, BlobPorter eller AzCopy.
Köra ett arbetsflöde via Microsoft Genomics-tjänsten med Hjälp av msgen Python-klienten
Om du vill köra ett arbetsflöde via Microsoft Genomics-tjänsten redigerar duconfig.txt-filen för att ange lagringscontainern för in- och utdata för dina data. Öppna filenconfig.txt som du laddade ned från ditt Genomics-konto. De avsnitt som du behöver ange är din prenumerationsnyckel och de sex objekten längst ned, lagringskontots namn, nyckel och containernamn för både indata och utdata. Du hittar den här informationen genom att navigera i Azure Portal till Åtkomstnycklar för ditt lagringskonto eller direkt från Azure Storage Explorer.
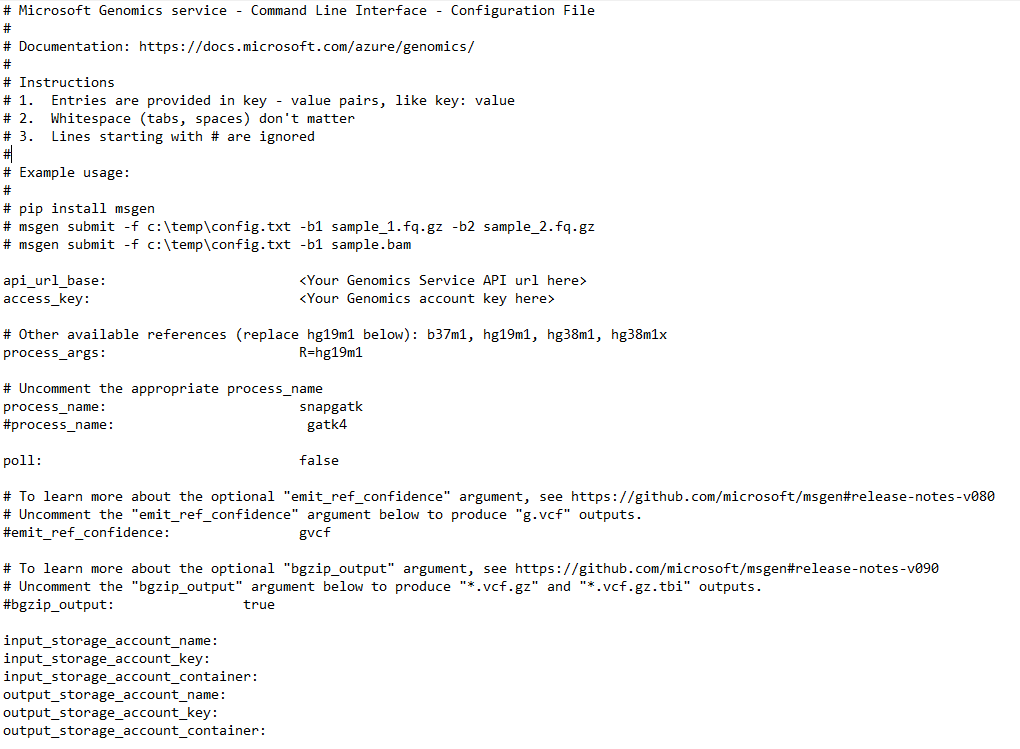
Om du vill köra GATK4 anger du parametern process_name till gatk4.
Som standard visar Genomics-tjänsten VCF-filer. Om du vill ha en gVCF-utdata i stället för VCF-utdata (motsvarande -emitRefConfidence i GATK 3.x och emit-ref-confidence i GATK 4.x) lägger du till parametern emit_ref_confidence i dinconfig.txt och anger den till gvcf, enligt föregående bild. Om du vill ändra tillbaka till VCF-utdata tar du antingen bort den från config.txt-filen eller anger parametern emit_ref_confidence till none.
bgzip är ett verktyg som komprimerar vcf- eller gvcf-filen och tabix skapar ett index för den komprimerade filen. Som standard körs bgzip Genomics-tjänsten följt av tabix ".g.vcf"-utdata men kör inte dessa verktyg som standard för ".vcf"-utdata. När tjänsten körs skapas filerna ".gz" (bgzip output) och ".tbi" (tabix output). Argumentet är ett booleskt värde, som är inställt på false som standard för ".vcf"-utdata och till true som standard för ".g.vcf"-utdata. Om du vill använda på kommandoraden anger -bz du eller --bgzip-output som true (kör bgzip och tabix) eller false. Om du vill använda det här argumentet i filenconfig.txt lägger du till bgzip_output: true eller bgzip_output: false i filen.
Skicka arbetsflödet till Microsoft Genomics-tjänsten med Hjälp av msgen Python-klienten
Använd Microsoft Genomics Python-klienten för att skicka ditt arbetsflöde med följande kommando:
msgen submit -f [full path to your config file] -b1 [name of your first paired end read] -b2 [name of your second paired end read]
Du kan visa statusen för dina arbetsflöden med hjälp av följande kommando:
msgen list -f c:\temp\config.txt
När arbetsflödet är klart kan du visa utdatafilerna i ditt Azure Storage-konto i den utdatacontainer som du har konfigurerat.
Nästa steg
I den här artikeln laddade du upp exempeldata till Azure Storage och skickade ett arbetsflöde till Microsoft Genomics-tjänsten via msgen Python-klienten. Mer information om andra indatafiltyper som kan användas med Microsoft Genomics-tjänsten finns på följande sidor: parade FASTQ | BAM | Multiple FASTQ eller BAM.
Feedback
Kommer snart: Under hela 2024 kommer vi att fasa ut GitHub-problem som feedbackmekanism för innehåll och ersätta det med ett nytt feedbacksystem. Mer information finns i: https://aka.ms/ContentUserFeedback.
Skicka och visa feedback för