Démarrage rapide : Exécuter un workflow par le biais du service Microsoft Genomics
Dans ce guide de démarrage rapide, vous chargez des données d’entrée sur un compte de stockage Blob Azure et exécutez un workflow par le biais du service Microsoft Genomics à l’aide du client Genomics Python. Microsoft Genomics est un service sécurisé et évolutif dédié à l’analyse secondaire de génomes. Il procède rapidement, en commençant par des lectures brutes, puis produit des lectures alignées et des appels de variants.
Prérequis
- Compte Azure avec un abonnement actif. Créez un compte gratuitement.
- Python 2.7.12+, avec
pipinstallé, etpythondans le chemin de votre système. Le client Microsoft Genomics n’est pas compatible avec Python 3.
Configuration : Créer un compte Microsoft Genomics dans le portail Azure
Pour créer un compte Microsoft Genomics, accédez à Créer un compte Genomics dans le portail Azure. Si vous n’avez pas encore d’abonnement Azure, créez-en un avant de créer le compte Microsoft Genomics.
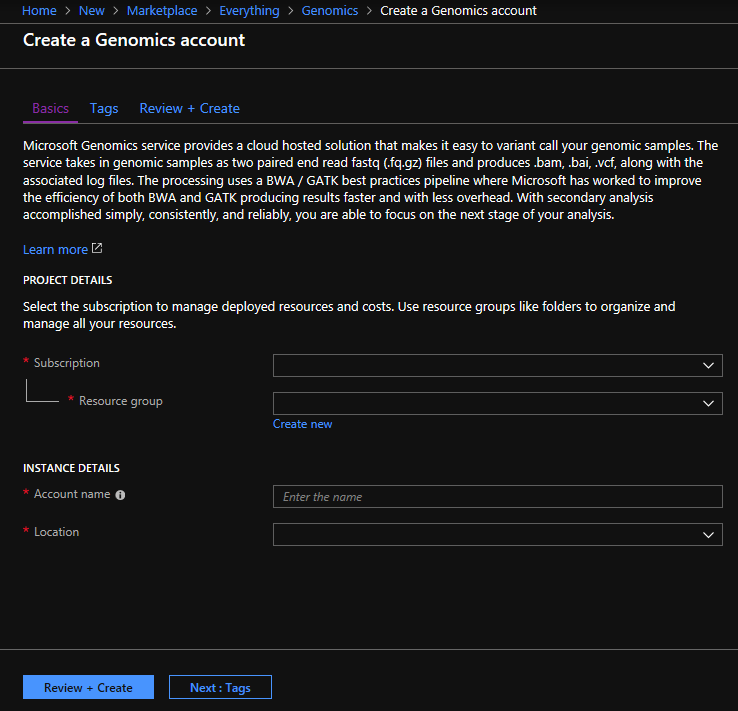
Configurez votre compte Genomics avec les informations suivantes, tel que représenté dans l’image précédente.
| Paramètre | Valeur suggérée | Description du champ |
|---|---|---|
| Abonnement | Votre nom d’abonnement | Il s’agit de l’unité de facturation de vos services Azure. Pour plus de détails sur votre abonnement, consultez la section Abonnements |
| Resource group | myResourceGroup | Les groupes de ressources vous permettent de rassembler plusieurs ressources Azure (compte de stockage, compte Genomics, etc.) dans un groupe unique, pour une gestion simplifiée. Pour plus d’informations, consultez Groupes de ressources. Pour connaître les noms valides de groupes de ressources, consultez la page Règles d’affectation des noms. |
| Nom du compte | MyGenomicsAccount | Choisissez un identifiant de compte unique. Pour connaître les noms valides, consultez la page Règles d’affectation des noms. |
| Emplacement | USA Ouest 2 | Le service est disponible dans les régions USA Ouest 2, Europe Ouest et Asie Sud-Est. |
Pour superviser le processus de déploiement, vous pouvez sélectionner les Notifications dans la barre de menus supérieure.

Pour plus d’informations sur Microsoft Genomics, consultez la page Qu’est-ce que Microsoft Genomics ?
Configuration : Installer le client Microsoft Genomics Python
Vous devez installer Python et le client Microsoft Genomics Python msgen dans votre environnement local.
Installation de Python
Le client Microsoft Genomics Python est compatible avec Python 2.7.12 ou une version 2.7.xx ultérieure. 2.7.14 est la version suggérée. Accédez au téléchargement ici.
Important
Python 3.x n’est pas compatible avec Python 2.7.xx. msgen est une application Python 2.7. Quand vous exécutez msgen, vérifiez que votre environnement Python actif utilise une version 2.7.xx de Python. Vous pouvez obtenir des erreurs si vous tentez d’utiliser msgen avec une version 3.x de Python.
Installer le client Microsoft Genomics Pythonmsgen
Utilisez Python pip pour installer le client Microsoft Genomics msgen. Les instructions suivantes supposent que Python 2.x est déjà dans votre chemin système. Si l’installation de pip n’est pas reconnue, vous devez ajouter Python et le sous-dossier des scripts à votre chemin système.
pip install --upgrade --no-deps msgen
pip install msgen
Si vous ne souhaitez pas installer msgen en tant qu’instance binaire au sein du système et modifier les packages Python à l’échelle du système, utilisez la balise –-user avec pip.
Quand vous utilisez l’installation basée sur le package ou setup.py, tous les packages nécessaires sont installés.
Tester le client Python msgen
Pour tester le client Microsoft Genomics, téléchargez le fichier de configuration à partir du compte Genomics. Dans le portail Azure, accédez à votre compte Genomics en sélectionnant Tous les services dans la zone supérieure gauche, puis en recherchant et en sélectionnant les comptes Genomics.
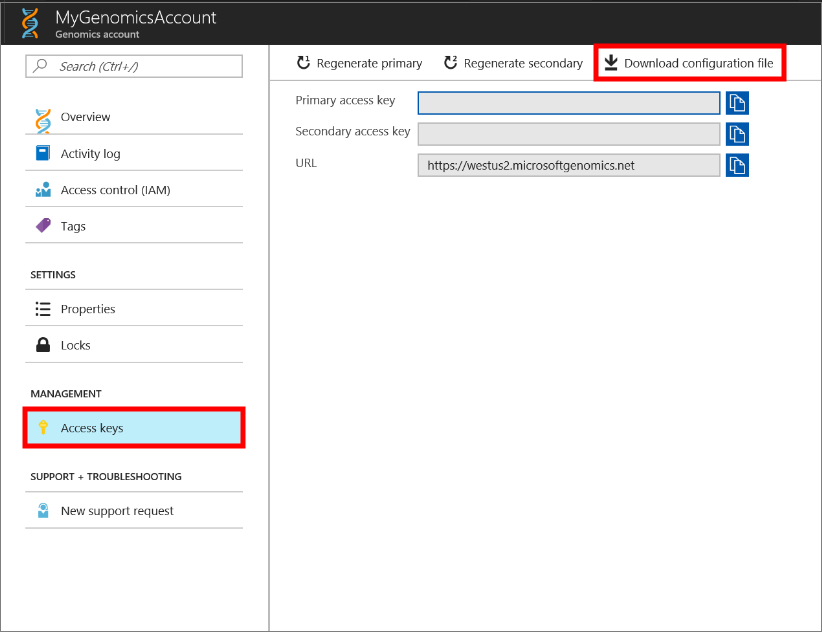
Sélectionnez le compte Genomics créé, accédez à Clés d’accès, puis téléchargez le fichier de configuration.
Vérifiez que le client Microsoft Genomics Python fonctionne avec la commande suivante :
msgen list -f "<full path where you saved the config file>"
Créer un compte Microsoft Stockage Azure
Le service Microsoft Genomics suppose que les entrées sont stockées en tant qu’objets blob de bloc dans un compte Stockage Azure. Il écrit également les fichiers de sortie en tant qu’objets blob de bloc sur un conteneur spécifié par l’utilisateur, dans un compte Stockage Azure. Les entrées et les sorties peuvent être hébergées dans différents comptes de stockage. Si vous avez déjà déplacé vos données dans un compte Stockage Azure, il vous suffit simplement de vous assurer qu’elles sont hébergées au même emplacement que votre compte Genomics. Dans le cas contraire, des frais de sortie s’appliquent lors de l’exécution du service Microsoft Genomics. Si vous ne possédez pas encore de compte de stockage Azure, vous devez en créer un, dans lequel vous chargez vos données. Vous trouverez plus d’informations sur les comptes de stockage Azure ici. Cette section vous donne accès à la définition d’un compte de stockage et à la description des services fournis. Pour créer un compte de stockage Azure, accédez à Créer un compte de stockage dans le portail Azure.
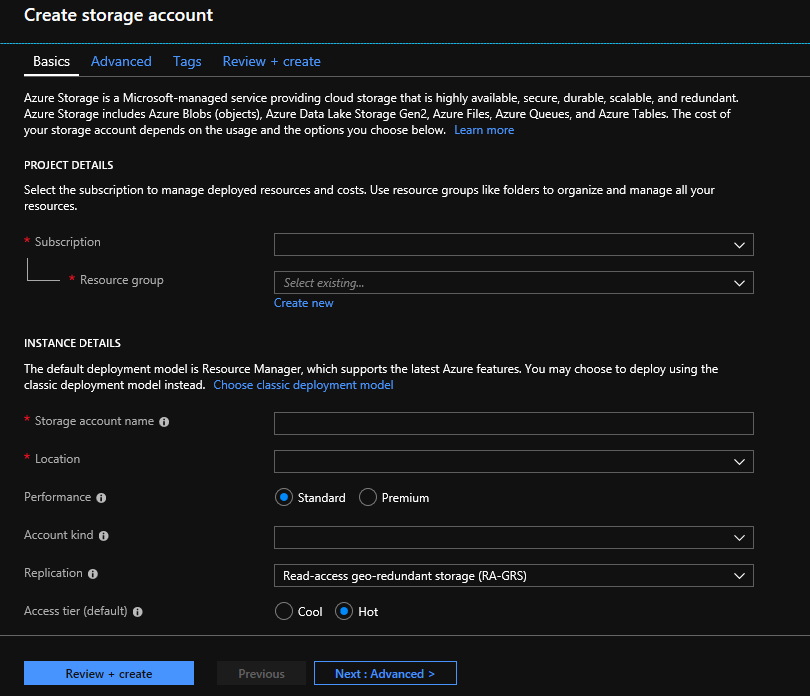
Configurez votre compte de stockage avec les informations suivantes, tel que représenté dans l’image précédente. Utilisez la plupart des options standard dédiées aux comptes de stockage, en indiquant uniquement qu’il s’agit d’un compte de stockage BlobStorage, non universel. Le stockage d’objets blob peut s’avérer entre 2 à 5 fois plus rapide pour les téléchargements et les chargements. Le modèle de déploiement par défaut, Azure Resource Manager, est recommandé.
| Paramètre | Valeur suggérée | Description du champ |
|---|---|---|
| Abonnement | Votre abonnement Azure | Pour plus d’informations sur votre abonnement, consultez Abonnements. |
| Resource group | myResourceGroup | Vous pouvez sélectionner le même groupe de ressources en tant que compte Genomics. Pour connaître les noms valides de groupes de ressources, consultez la page Règles d’affectation des noms. |
| Nom du compte de stockage | MyStorageAccount | Choisissez un identifiant de compte unique. Pour connaître les noms valides, consultez la page Règles d’affectation des noms. |
| Emplacement | USA Ouest 2 | Utilisez l’emplacement de votre compte Genomics, afin de réduire les frais de sortie ainsi que la latence. |
| Performances | Standard | Le niveau par défaut est Standard. Pour plus d’informations sur les comptes de stockage standard et premium, consultez Présentation du Stockage Microsoft Azure. |
| Type de compte | BlobStorage | Le stockage d’objets blob peut s’avérer entre 2 à 5 fois plus rapide que le modèle à usage général pour les téléchargements et les chargements. |
| Réplication | Stockage localement redondant | Le stockage localement redondant réplique vos données dans la région du centre de données dans laquelle vous avez créé votre compte de stockage. Pour plus d’informations, consultez l’article Réplication de Stockage Azure. |
| Niveau d’accès | À chaud | Un niveau d’accès chaud indique que le compte de stockage sera sollicité plus fréquemment. |
Ensuite, sélectionnez Vérifier + créer pour créer votre compte de stockage. Comme vous l’avez fait pour la création de votre compte Genomics, vous pouvez sélectionner les Notifications dans la barre de menus supérieure pour superviser le processus de déploiement.
Charger les données d’entrée dans votre compte de stockage
Le service Microsoft Genomics prend en charge des séquences appariées (fichiers fastq ou bam) en tant que fichiers d’entrée. Vous pouvez décider de charger vos propres données ou d’accéder aux exemples de données publiques mis à votre disposition.
Au sein de votre compte de stockage, vous devez créer un conteneur d’objets blob pour vos données d’entrée et un second conteneur d’objets blob pour vos données de sortie. Chargez les données d’entrée dans votre conteneur d’objets blob dédié. Pour ce faire, divers outils sont à votre disposition, notamment Explorateur Stockage Microsoft Azure, BlobPorter ou AzCopy.
Exécuter un workflow via le service Microsoft Genomics en utilisant le client Python msgen
Pour exécuter un workflow via le service Microsoft Genomics, modifiez le fichier config.txt afin de spécifier les conteneurs de stockage d’entrée et de sortie dédiés à vos données. Ouvrez le fichier config.txt téléchargé à partir de votre compte Genomics. Les sections à spécifier sont votre clé d’abonnement et les six éléments inférieurs, le nom du compte de stockage, la clé et les noms des conteneurs d’entrée et de sortie. Vous pouvez trouver ces informations en accédant au portail Azure des Clés d’accès à votre compte de stockage, ou directement à partir de l’Explorateur Stockage Microsoft Azure.
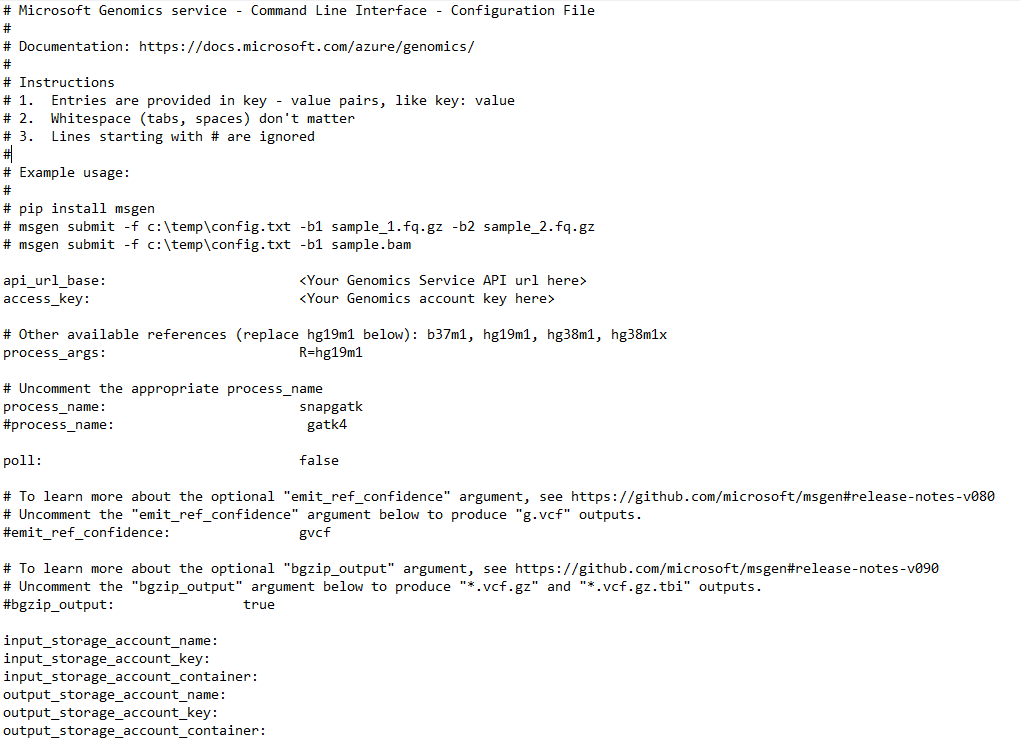
Si vous souhaitez exécuter GATK4, définissez le paramètre process_name sur gatk4.
Par défaut, le service Genomics génère des fichiers VCF. Si vous voulez une sortie gVCF plutôt qu’une sortie VCF (équivalent à -emitRefConfidence dans GATK 3.x et à emit-ref-confidence dans GATK 4.x), ajoutez le paramètre emit_ref_confidence à votre fichier config.txt, puis définissez-le sur gvcf, comme illustré dans la figure précédente. Pour revenir à la sortie VCF, supprimez-le du fichier config.txt ou définissez le paramètre emit_ref_confidence sur none.
bgzip est un outil qui compresse le fichier vcf ou gvcf et qui tabix crée un index pour le fichier compressé. Par défaut, le service Genomics exécute bgzip suivi de tabix sur la sortie « .g.vcf », mais il n’exécute pas ces outils par défaut pour la sortie « .vcf ». Lors de son exécution, le service génère des fichiers « .gz » (sortie bgzip) et « .tbi » (sortie tabix). L’argument est un booléen, qui est défini sur false par défaut pour la sortie « .vcf » et sur true par défaut pour la sortie « .g.vcf ». Pour l’utiliser sur la ligne de commande, spécifiez -bz ou --bgzip-output comme true (exécuter bgzip et tabix) ou false. Pour utiliser cet argument dans le fichier config.txt, ajoutez bgzip_output: true ou bgzip_output: false au fichier.
Soumettre votre workflow au service Microsoft Genomics en utilisant le client Python msgen
Utilisez le client Microsoft Genomics Python pour soumettre votre workflow à l’aide de la commande suivante :
msgen submit -f [full path to your config file] -b1 [name of your first paired end read] -b2 [name of your second paired end read]
Vous pouvez afficher l’état de votre workflow à l’aide de la commande suivante :
msgen list -f c:\temp\config.txt
Une fois le workflow terminé, vous avez accès aux fichiers de sortie dans votre compte de stockage Azure, dans le conteneur de sortie configuré.
Étapes suivantes
Dans cet article, vous avez chargé un exemple de données d’entrée dans le stockage Azure et soumis un workflow dans le service Microsoft Genomics, via le client Python msgen. Pour en savoir plus sur les autres types de fichiers d’entrée pouvant être utilisés avec le service Microsoft Genomics, consultez les pages suivantes : Soumettre un workflow à l’aide d’entrées de fichiers FASTQ | Soumettre un workflow à l’aide d’une entrée de fichier BAM | Soumettre un workflow à l’aide d’entrées multiples d’un seul exemple.