Snelstartgids: een werkstroom uitvoeren via de service Microsoft Genomics
In deze quickstart uploadt u invoergegevens naar een Azure Blob Storage-account en voert u een werkstroom uit via de Microsoft Genomics-service met behulp van de Python Genomics-client. Microsoft Genomics is een schaalbare, veilige service voor secundaire analyse waarbij genomen snel kunnen worden verwerkt. Hierbij wordt begonnen met onbewerkte leesbewerkingen en worden vervolgens uitgelijnde leesbewerkingen en variant-aanroepen geproduceerd.
Vereisten
- Een Azure-account met een actief abonnement. Gratis een account maken
- Python 2.7.12 + met
pipgeïnstalleerd enpythonin het systeempad. De Microsoft Genomics-client is niet compatibel met Python 3.
Instellen: een Microsoft Genomics-account in Azure Portal maken
Ga voor het maken van een Microsoft Genomics-account naar Een Genomics-account maken in Azure Portal. Als u nog geen Azure-abonnement hebt, maakt u er een voordat u een Microsoft Genomics-account maakt.
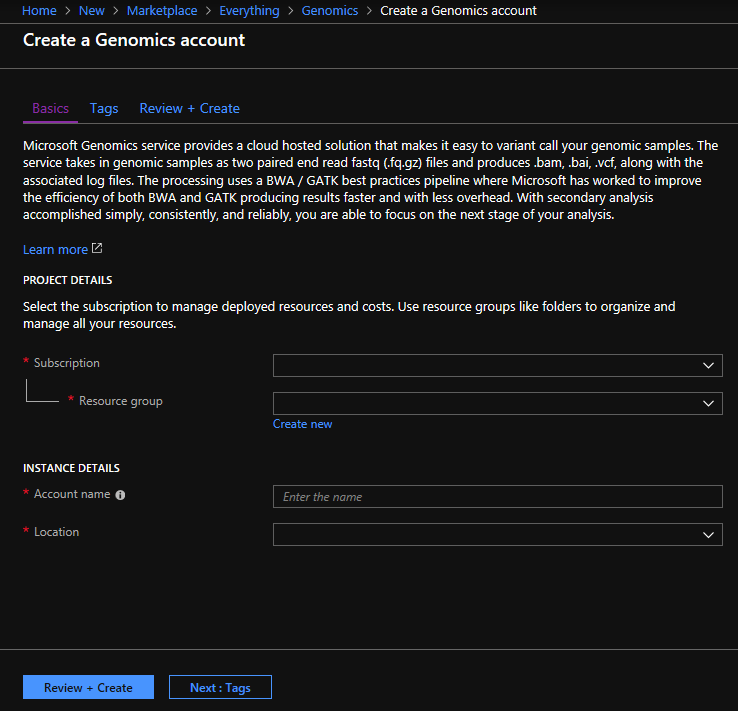
Configureer uw Genomics-account met de volgende informatie, zoals weergegeven in de vorige afbeelding.
| Instelling | Voorgestelde waarde | Veldomschrijving |
|---|---|---|
| Abonnement | De naam van uw abonnement | Dit is de factureringseenheid voor uw Azure-services; zie Abonnementen voor meer informatie over uw abonnement. |
| Resourcegroep | MyResourceGroup | Met resourcegroepen kunt u meerdere Azure-resources (opslagaccount, Genomics-account enzovoort) in één groep indelen voor eenvoudig beheer. Zie Resourcegroepen voor meer informatie. Zie Naamgevingsregels voor geldige resourcegroepnamen. |
| Accountnaam | MyGenomicsAccount | Kies een unieke account-id. Zie Naamgevingsregels voor geldige namen. |
| Locatie | VS - west 2 | De service is beschikbaar in VS - west 2, Europa - west en Azië - zuidoost |
U kunt in de bovenste menubalk Meldingen selecteren om het implementatieproces te controleren.

Zie Wat is Microsoft Genomics? voor meer informatie over Microsoft Genomics.
Instellen: de Python-client voor Microsoft Genomics installeren
U moet zowel Python als de Python-client voor Microsoft Genomics msgen in uw lokale omgeving installeren.
Python installeren
De Python-client voor Microsoft Genomics is compatibel met Python 2.7.12 of een nieuwere 2.7.xx-versie. 2.7.14 is de voorgestelde versie. U vindt de download hier.
Belangrijk
Python 3.x is niet compatibel met Python 2.7.xx. msgen is een Python 2.7-app. Zorg er bij het uitvoeren van msgen voor dat uw actieve Python-omgeving een 2.7.xx-versie van Python gebruikt. Er treden mogelijk fouten op bij het gebruik van msgen met een 3.x-versie van Python.
De Python-client voor Microsoft Genomics msgen installeren
Gebruik Python-pip om de Microsoft Genomics-client msgen te installeren. Bij de volgende instructies wordt ervan uitgegaan dat Python 2.x al in het systeempad staat. Als de installatie van pip niet wordt herkend, moet u Python en de submap voor scripts aan het systeempad toe te voegen.
pip install --upgrade --no-deps msgen
pip install msgen
Als u msgen niet wilt installeren als een systeembreed binair bestand en systeembrede Python-pakketten niet wilt wijzigen, gebruikt u de markering –-user met pip.
Als u de installatie op basis van een pakket of setup.py gebruikt, worden alle vereiste pakketten geïnstalleerd.
Test de Python-client msgen
Download het configuratiebestand in uw Genomics-account om de client voor Microsoft Genomics te testen. Navigeer in Azure Portal naar uw Genomics-account door links bovenin Alle services te selecteren en vervolgens Genomics-accounts te zoeken en te selecteren.
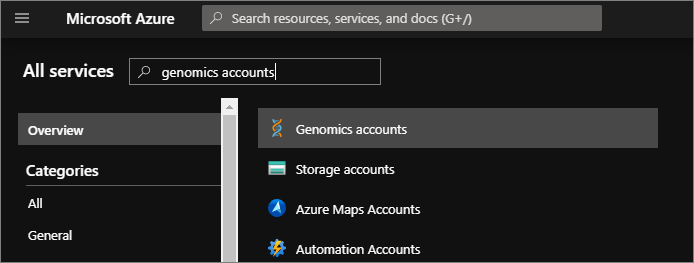
Selecteer het Genomics-account dat u zojuist hebt gemaakt, ga naar Toegangssleutels en download het configuratiebestand.
Test of de Python-client voor Microsoft Genomics werkt met de volgende opdracht
msgen list -f "<full path where you saved the config file>"
Een Microsoft Azure Storage-account maken
De service Microsoft Genomics verwacht dat invoeren worden opgeslagen als blok-blobs in een Azure-opslagaccount. Ook schrijft de service uitvoerbestanden als blok-blobs naar een door de gebruiker opgegeven container in een Azure-opslagaccount. De invoeren en uitvoeren kunnen zich in verschillende opslagaccounts bevinden. Als u uw gegevens al in een Azure-opslagaccount hebt, hoeft u alleen ervoor te zorgen dat deze zich op dezelfde locatie bevinden als uw Genomics-account. Anders worden er uitvoerkosten gemaakt wanneer de Microsoft Genomics-service wordt uitgevoerd. Als u nog geen Azure-opslagaccount hebt, dient u er een te maken en uw gegevens te uploaden. U vindt hier meer informatie over Azure-opslagaccounts, met inbegrip van wat een opslagaccount is en welke services het biedt. Ga naar Opslagaccount maken in Azure Portal om een Azure-opslagaccount te maken.
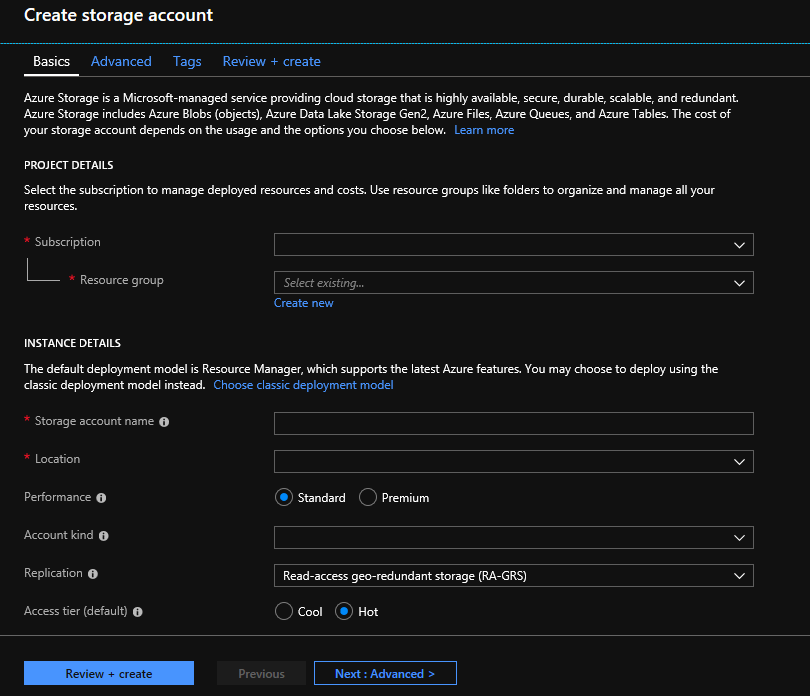
Configureer uw opslagaccount met de volgende informatie, zoals weergegeven in de vorige afbeelding. Gebruik de meeste van de standaardopties voor een opslagaccount en geef alleen op dat het account BlobStorage is en niet algemeen. Blob-opslag kan twee tot vijf keer sneller zijn voor downloads en uploads. Het standaardimplementatiemodel, Azure Resource Manager, wordt aanbevolen.
| Instelling | Voorgestelde waarde | Veldomschrijving |
|---|---|---|
| Abonnement | Uw Azure-abonnement | Zie Abonnementen voor meer informatie over uw abonnement. |
| Resourcegroep | MyResourceGroup | U kunt dezelfde resourcegroep als voor uw Genomics-account selecteren. Zie Naamgevingsregels voor geldige resourcegroepnamen |
| Naam van het opslagaccount | MyStorageAccount | Kies een unieke account-id. Zie Naamgevingsregels voor geldige namen |
| Locatie | VS - west 2 | Gebruik dezelfde locatie als de locatie van uw Genomics-account om uitvoerkosten te verminderen en latentie te beperken. |
| Prestaties | Standaard | Standard is de standaardinstelling. Zie Kennismaking met Microsoft Azure Storage voor meer informatie over standaard- en premium-opslagaccounts |
| Soort account | BlobStorage | Blob-opslag kan twee tot vijf keer sneller zijn dan algemeen gebruik voor downloads en uploads. |
| Replicatie | Lokaal redundante opslag | Lokaal redundante opslag repliceert uw gegevens in het datacenter in de regio waarin u uw opslagaccount hebt gemaakt. Zie Azure Storage-replicatie voor meer informatie. |
| Toegangslaag | Heet | De toegangslaag Hot geeft aan dat de objecten in het opslagaccount vaker worden gebruikt. |
Selecteer daarna Beoordelen en maken om uw opslagaccount te maken. Net als bij het maken van uw Genomics-account, kunt u in de bovenste menubalk Meldingen selecteren om het implementatieproces te controleren.
De invoergegevens naar uw opslagaccount uploaden
De Microsoft Genomics-service verwacht leesbewerkingen voor eindsequenties (FASTQ- of BAM-bestanden) als invoerbestanden. U kunt uw eigen gegevens uploaden, of openbare voorbeeldgegevens verkennen die voor u beschikbaar zijn gesteld.
U dient binnen uw opslagaccount een blob-container te maken voor uw invoergegevens en een tweede blob-container voor de uitvoergegevens. Upload de invoergegevens naar uw blob-container voor invoer. U kunt hiervoor verschillende hulpprogramma's gebruiken, waaronder Microsoft Azure Storage Explorer, BlobPorter en AzCopy.
Een werkstroom uitvoeren via de Microsoft Genomics-service met behulp van de Python-client msgen
Als u een werkstroom wilt uitvoeren via de service Microsoft Genomics, bewerkt u het bestand config.txt om de invoer- en uitvoeropslagcontainer voor uw gegevens op te geven. Open het bestand config.txt dat u van uw Genomics-account hebt gedownload. De gedeelten die u dient op te geven zijn de abonnementssleutel en de zes items onderaan, de naam van het opslagaccount, de sleutel en de naam van de container voor zowel invoer als uitvoer. U kunt deze informatie vinden door in Azure Portal naar Toegangssleutels voor uw opslagaccount te navigeren of rechtstreeks vanuit Azure Storage Explorer.
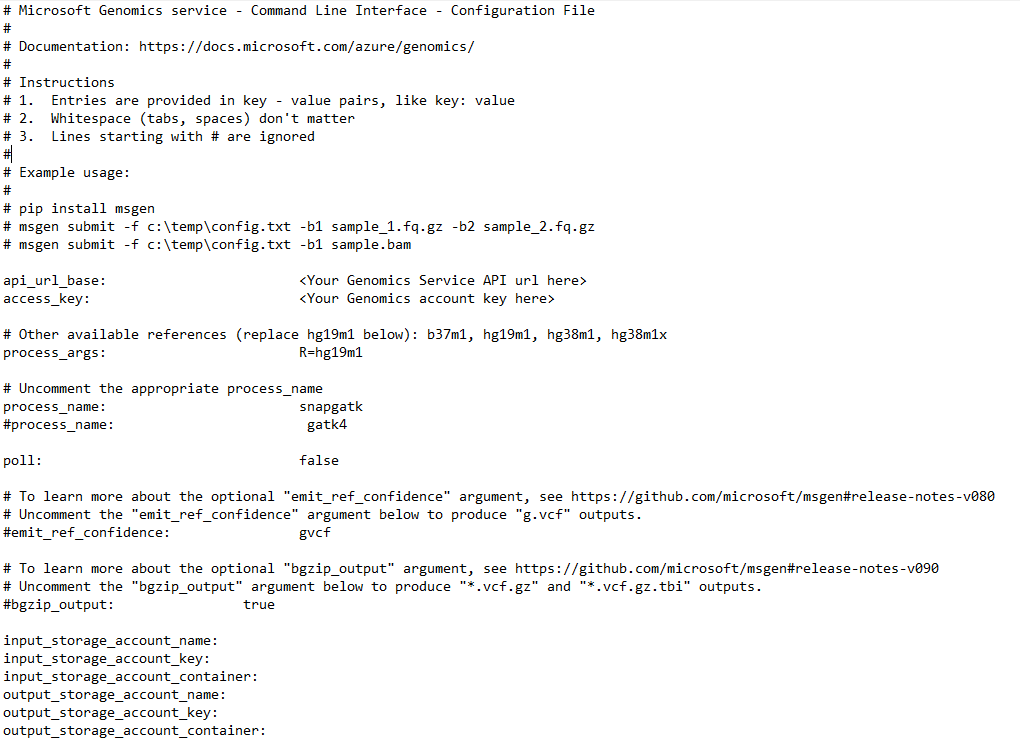
Als u graag GATK4 wilt uitvoeren, stelt u de parameter process_name in op gatk4.
De Genomics-service voert standaard VCF-bestanden uit. Als u graag gVCF-uitvoer wilt in plaats van VCF-uitvoer (gelijk aan -emitRefConfidence in GATK 3.x en emit-ref-confidence in GATK 4.x), voegt u de parameter emit_ref_confidence toe aan uw config.txt en stelt u deze in op gvcf, zoals wordt weergegeven in de vorige afbeelding. Als u weer terug wilt naar VCF-uitvoer, verwijdert u deze uit het bestand config.txt of stelt u de parameter emit_ref_confidence in op none.
bgzip is een hulpprogramma waarmee het VCF- of gVCF-bestand wordt gecomprimeerd en tabix een index voor het gecomprimeerde bestand maakt. De Genomics-service voert standaard bgzip gevolgd door tabix uit voor '.g.vcf'-uitvoer, maar voert deze hulpprogramma's niet standaard uit voor '.vcf '-uitvoer. Wanneer u de service uitvoert, resulteert dit in '.gz'- (bgzip-uitvoer) en 'tbi'-bestanden (tabix-uitvoer). Het argument is een Booleaanse waarde, die standaard is ingesteld op False voor '.vcf'-uitvoer en standaard is ingesteld op True voor '.g.vcf'-uitvoer. Als u dit wilt gebruiken op de opdrachtregel, geeft u -bz of --bgzip-output op als true (voer bgzip en tabix uit) of false. Als u dit argument wilt gebruiken in het bestand config.txt, voegt u bgzip_output: true of bgzip_output: false toe aan het bestand.
Uw werkstroom verzenden naar de Microsoft Genomics-service met behulp van de Python-client msgen
Gebruik de Python-client voor Microsoft Genomics om uw werkstroom te verzenden met de volgende opdracht:
msgen submit -f [full path to your config file] -b1 [name of your first paired end read] -b2 [name of your second paired end read]
U kunt de status van uw werkstromen bekijken met de volgende opdracht:
msgen list -f c:\temp\config.txt
Nadat de werkstroom is voltooid, kunt u de uitvoerbestanden weergeven in uw Azure-opslagaccount in de uitvoercontainer die u hebt geconfigureerd.
Volgende stappen
In dit artikel hebt u voorbeeldinvoergegevens geüpload naar Azure Storage en een werkstroom verzonden naar de Microsoft Genomics-service via de Python-client voor msgen. Ga voor meer informatie over andere invoerbestandstypen die kunnen worden gebruikt met de Microsoft Genomics-service naar de volgende pagina's: paired FASTQ | BAM | Multiple FASTQ or BAM (gekoppelde FASTQ/BAM/Meerdere FASTQ of BAM).