Déployer et configurer l’ingestion des données DICOM dans les solutions de données de santé (version préliminaire)
[Cet article fait partie de la documentation en version préliminaire et peut faire l’objet de modifications.]
L’Ingestion des données DICOM vous permet d’utiliser le pipeline d’ingestion des données d’imagerie et d’importer vos données DICOM (Digital Imaging and Communications in Medicine) dans OneLake. Vous pouvez déployer et configurer la fonctionnalité après avoir déployé les solutions de données de santé (version préliminaire) et la fonctionnalité Sources des données de santé sur votre espace de travail Fabric.
L’ingestion des données DICOM est une fonctionnalité facultative dans les solutions de données de santé dans Microsoft Fabric (version préliminaire). Vous avez la possibilité de décider de l’utiliser ou non, en fonction de vos besoins ou scénarios spécifiques.
Conditions préalables au déploiement
Déployez les solutions de données de santé (version préliminaire) dans votre espace de travail Fabric.
Note
Pour la fonctionnalité d’imagerie d’ingestion des données DICOM, il n’est pas nécessaire de déployer les services de données de santé Azure (service FHIR), le service de langage Azure ou l’offre Place de marché Azure.
L’ingestion des données DICOM dépend directement de la fonctionnalité Sources des données de santé. Assurez-vous d’abord de déployer, de configurer et d’exécuter avec succès les pipelines des sources de données de santé. Pour plus d’informations, consultez Déployer et configurer les sources des données de santé.
Déployez et configurez la fonctionnalité d’analyse OMOP (facultatif).
Téléchargez les exemples de données dans votre environnement, comme expliqué dans Déployer les exemples de données. Si vous avez déjà téléchargé les exemples de données, vérifiez si votre téléchargement inclut les exemples de jeux de données d’imagerie DICOM.
Assurez-vous que la version d’exécution Fabric est définie sur Runtime 1.1 dans les paramètres de l’espace de travail. Pour plus d’informations, consultez Réinitialiser la version d’exécution Spark dans l’espace de travail Fabric.
Configurez et exécutez les notebooks suivants en fonction des exemples de données :
Après avoir terminé ces étapes, vous devriez avoir déployé toutes les lakehouses et les exemples d’ensembles de données cliniques ingérés dans les tables delta bronze, argent et or.
Pour vérifier le déploiement et l’ingestion des exemples d’ensembles de données :
- Confirmez si vous pouvez voir les trois lakehouses dans la fenêtre de votre espace de travail.
- Exécutez un exemple de requête sur la table care_site dans la lakehouse OMOP (or).
Déployer l’ingestion des données DICOM
Pour déployer la fonctionnalité d’ingestion des données DICOM sur votre espace de travail, procédez comme suit :
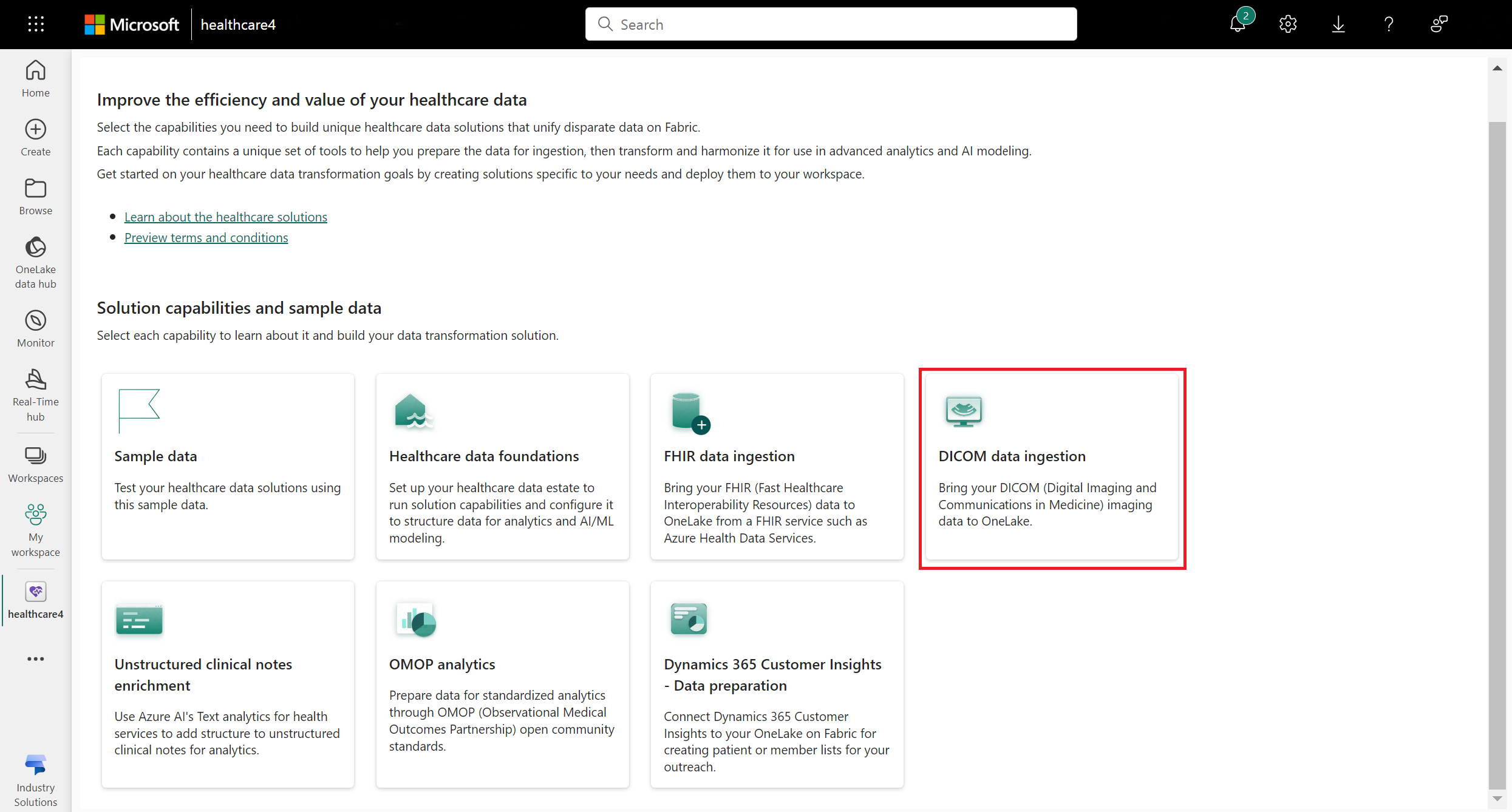
Accédez à la page d’accueil des solutions de données de santé sur Fabric.
Sélectionnez la vignette d’ingestion des données DICOM.
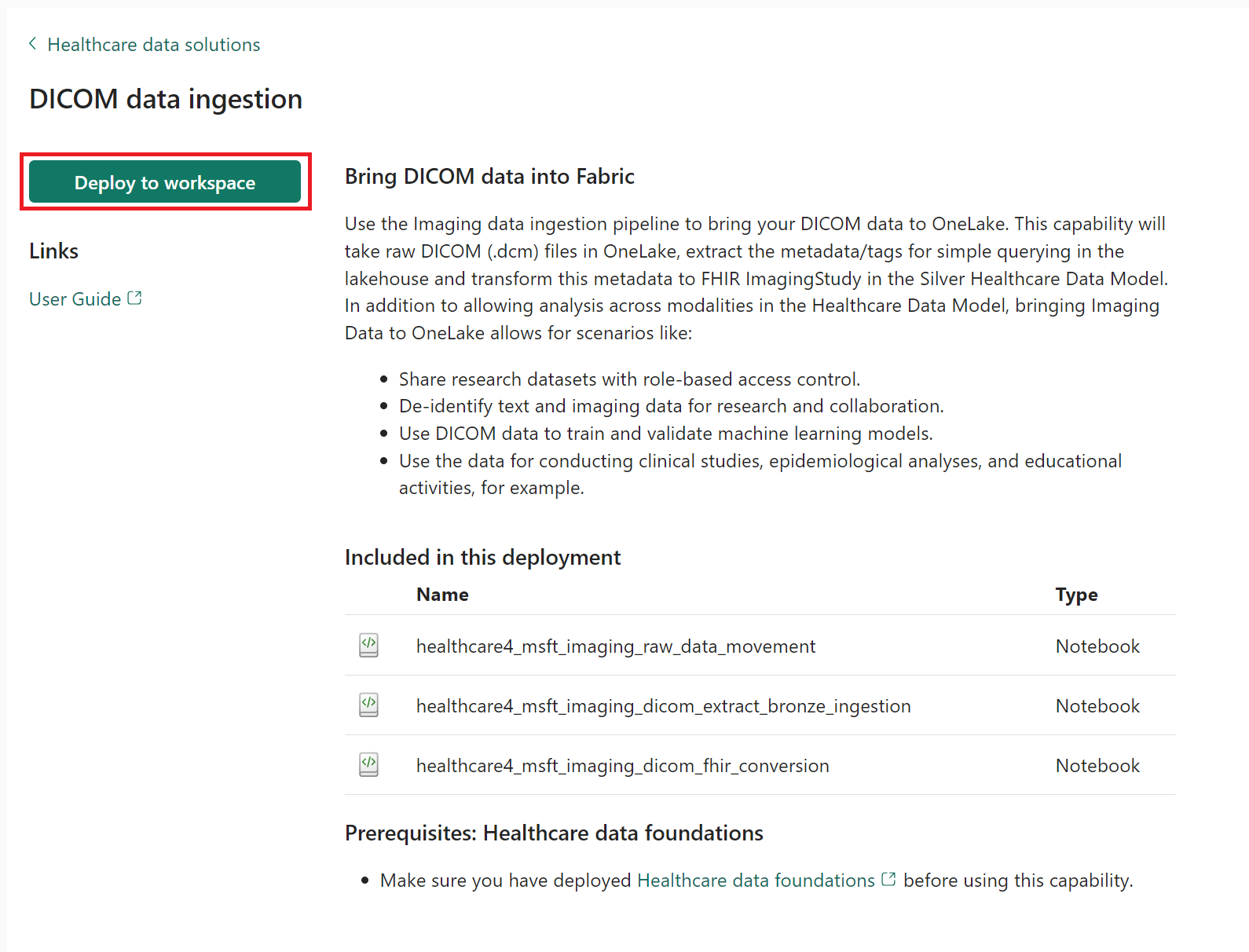
Sur la page des fonctionnalités, sélectionnez Déployer sur l’espace de travail.
Le déploiement peut prendre plusieurs minutes. Ne fermez pas l’onglet ou le navigateur pendant que le déploiement est en cours. Pendant que vous patientez, vous pouvez travailler dans un autre onglet.
Une fois le déploiement terminé, vous pouvez voir une notification dans la barre de messages.
Sélectionnez Gérer la capacité dans la barre de messages pour accéder à la page Gestion des capacités.
Ici, vous pouvez afficher, configurer et gérer les trois notebooks suivants déployés avec la fonctionnalité :
- healthcare#_msft_imaging_raw_data_movement
- healthcare#_msft_imaging_dicom_extract_bronze_ingestion
- healthcare#_msft_imaging_dicom_fhir_conversion
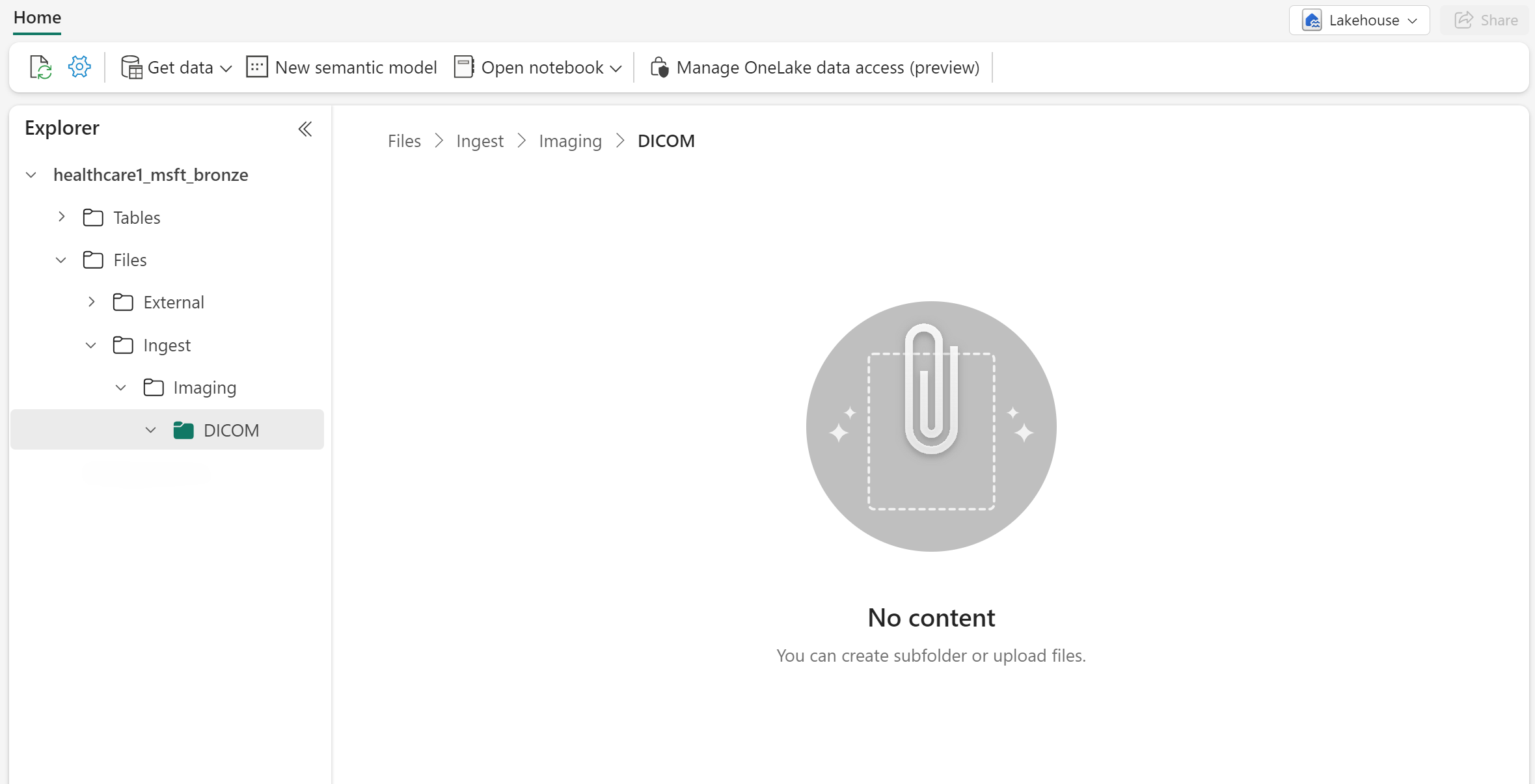
Dans le même environnement, accédez à la lakehouse healthcare#_msft_bronze et sélectionnez-la.
Sous Fichiers, créez trois sous-dossiers en utilisant le chemin de dossier suivant :
Files\Ingest\Imaging\DICOM.Le dossier Ingest devrait être vide maintenant. Il sert de dossier d’entrée pour ajouter les fichiers DICOM pour l’ingestion.
Notebooks d’ingestion des données DICOM
La fonctionnalité d’ingestion des données DICOM déploie les notebooks suivants dans votre environnement. Chaque notebook comprend un ensemble de paramètres configurables que vous pouvez examiner et modifier sur la page de configuration et de gestion de la configuration respective.
| Bloc-notes | Fonctionnalité |
|---|---|
| healthcare#_msft_imaging_raw_data_movement | Utilise le module ImagingRawDataMovementService de la bibliothèque de solutions de données de santé (version préliminaire) pour déplacer et extraire des fichiers d’imagerie médicale (fichiers DCM) d’un fichier ZIP. Les fichiers sont transférés d’un dossier de dépôt configurable vers une structure de dossiers appropriée en fonction de la date d’exécution (au format yyyy/mm/dd). |
| healthcare#_msft_imaging_dicom_extract_bronze_ingestion | Utilise le module MetadataExtractionOrchestrator de la bibliothèque de solutions de données de santé (version préliminaire) pour extraire les métadonnées DICOM des fichiers DCM. Les métadonnées sont ensuite stockées dans la table delta dicomimagingmetastore de la lakehouse bronze. |
| healthcare#_msft_imaging_dicom_fhir_conversion | Utilise le module MetadataToFhirConvertor de la bibliothèque de solutions de données de santé (version préliminaire) pour convertir les métadonnées DICOM dans la table delta bronze. Le processus de conversion implique la transformation des métadonnées de la table dicomimagingmetastore en ressource FHIR ImagingStudy au format FHIR R4.3. La sortie est enregistrée en tant que fichiers NDJSON. |
Exemples de données d’imagerie
Les exemples de données fournis avec les solutions de données de santé (version préliminaire) incluent les exemples d’ensembles de données d’imagerie que vous pouvez utiliser pour exécuter le pipeline d’ingestion des données DICOM. Vous pouvez également explorer la transformation et la progression des données à travers les lakehouses bronze, argent et or en médaillon. Les exemples de données d’imagerie fournis ne sont peut-être pas cliniquement significatifs, mais ils sont techniquement complets et détaillés pour démontrer les fonctionnalités d’imagerie de la solution. Pour accéder aux exemples de dossiers de données, assurez-vous de télécharger le dossier de données d’imagerie msft_dmh_imaging_data, comme expliqué dans Déployer les exemples de données.
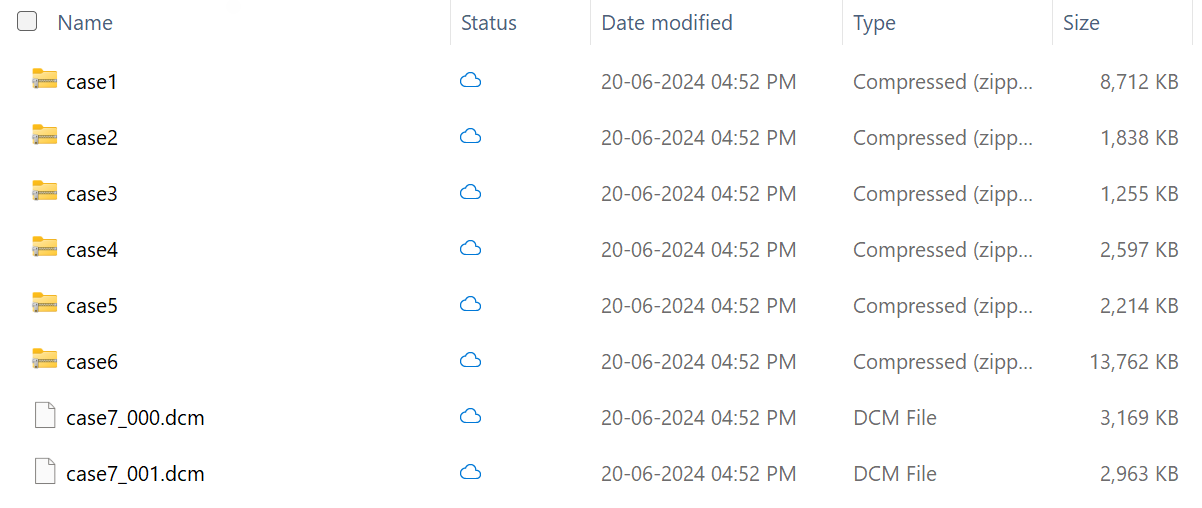
Dossier DICOM
Le dossier DICOM contient sept fichiers de cas. Les six premiers fichiers sont au format ZIP, tandis que le dernier fichier est au format DCM natif. Cette différence illustre la capacité du pipeline d’ingestion à ingérer des données DICOM aux formats compressés et natifs. Les identifiants des patients dans ces dossiers de cas d’imagerie correspondent aux patients dans les exemples de données cliniques.
Vous pouvez utiliser l’Explorateur de fichiers OneLake ou l’Explorateur de stockage Azure pour accéder aux exemples de données déployés.
Voici un aperçu détaillé montrant le nombre total d’études (9), de séries (24) et d’instances (96) dans le jeu de données d’imagerie :
| Fichier de cas | Description de l’étude (StudyInstanceUID) | Séries (modalités) | Instances |
|---|---|---|---|
| case1.zip | MRT Sakroiliakalgelenke (1.2.276.0.50.192168001099.7810872.14547392.270) | 1 (MR) | 31 |
| case2.zip | CT Abdomen (1.2.276.0.50.192168001099.8252157.14547392.4) | 2 (CT) | 7 |
| case3.zip | MRT Oberbauch (1.2.276.0.50.192168001092.11156604.14547392.4) | 5 (MR) | 7 |
| case4.zip | MRT Schädel (1.2.276.0.50.192168001099.8687553.14547392.4) | 4 (MR) | 30 |
| case5.zip | MRA (1.2.276.0.50.192168001092.11517584.14547392.4) MRT Oberbauch (1.2.276.0.50.192168001099.8829267.14547392.4) | 1 (OT) 5 (MR) | 5 6 |
| case6.zip | Thorax digital (1.2.276.0.50.192168001099.9140875.14547392.277) CT Thorax (1.2.276.0.50.192168001099.9140875.14547392.4) | 2 (CR) 2 (CT) | 2 6 |
| case7.dcm | Ellenbogen (1.2.276.0.50.192168001099.9483698.14547392.4) | 2 (CR) | 2 |
| Total | 9 | 24 | 96 |
où :
- CR = Radiographie informatisée
- CT = Tomodensitométrie
- MR/MRT = Tomographie par résonance magnétique, communément appelée Imagerie par résonance magnétique (IRM)
- ARM = Angiographie par résonance magnétique
- OT = Tomographie optique
Note
Certaines descriptions d’études dans le tableau précédent sont en allemand puisque les métadonnées de l’exemple du jeu de données d’imagerie sont en allemand. Voici les traductions françaises correspondantes pour ces termes:
| Allemand | Français |
|---|---|
| Ellenbogen | Coude |
| Oberbauch | Abdomen supérieur |
| Sakroiliakalgelenke | Articulations sacro-iliaques |
| Schädel | Crâne |
Dossier FHIR
Important
Il existe un problème connu avec les rapports de diagnostic dans les exemples de données. Les rapports de diagnostic sont actuellement déployés au format JSON au lieu du format NDJSON. Vous ne pouvez pas ingérer ces fichiers tels quels ; ils doivent d’abord être convertis au format NDJSON. Nous travaillerons à la résolution de ce problème dans la prochaine version.
Le dossier FHIR comprend six fichiers NDJSON contenant des exemples de ressources FHIR DiagnosticReport pour les six premiers fichiers de cas DICOM et leurs patients correspondants. Vous n’avez pas besoin d’ingérer ces rapports de diagnostic pour exécuter le pipeline d’ingestion DICOM, car les rapports sont considérés comme des données cliniques et sont ingérés dans le cadre de l’ingestion des données FHIR. Ce cas d’utilisation démontre les capacités multimodèles des solutions de données de santé (version préliminaire) qui présentent la capacité d’ingérer à la fois des données cliniques et d’imagerie, ce qui offre un scénario réaliste et cohérent.
Déployer l’API DICOM dans les services de données de santé Azure
Important
Suivez cette section de déploiement uniquement si vous utilisez le service DICOM dans les services de données de santé Azure. Sinon, vous pouvez ignorer cette section et passer à Configurer l’ingestion des données DICOM.
Les Services de données de santé Azure sont une solution basée sur le cloud qui vous aide à collecter, stocker et analyser des données de santé provenant de différentes sources et de différents formats. Ils prennent en charge diverses normes de santé, telles que DICOM. Le Service DICOM (qui fait partie des services de données de santé Azure) est une solution basée sur le cloud qui permet aux organismes de santé de stocker, gérer et échanger des données d’imagerie médicale de manière sécurisée et efficace avec n’importe quel système ou application DICOM compatible avec le web.
L’ingestion des données DICOM dans les solutions de données de santé (version préliminaire) dispose d’une intégration native avec le service DICOM. Si vous utilisez déjà le service DICOM, vous pouvez également utiliser les fonctionnalités d’analyse d’imagerie des solutions de données de santé (version préliminaire). Cette intégration native élimine la nécessité de l’intégration manuelle des ensembles de données entre les deux services. L’intégration est basée sur la fourniture d’un raccourci OneLake vers Azure Data Lake Storage Gen2 pour le service DICOM des services de données de santé Azure. Pour configurer l’intégration du lac de données, suivez les étapes décrites dans Déployer le service DICOM avec Azure Data Lake Storage. Pour plus d’informations sur ce pipeline d’ingestion, accédez à Option 3 : Intégration de bout en bout avec le service DICOM.
Configurer l’ingestion des données DICOM
La configuration de la fonctionnalité d’ingestion des données DICOM implique deux niveaux de configuration dans votre environnement de solutions de données de santé (version préliminaire) : la configuration globale et la configuration au niveau du notebook.
Configuration globale
Vous pouvez configurer le notebook healthcare#_msft_config_notebook pour configurer et gérer la configuration nécessaire pour toutes les transformations de données dans votre environnement de solutions de données de santé (version préliminaire). Pour plus d’informations, accédez à Configurer le notebook de configuration globale.
Configuration au niveau du notebook
La fonctionnalité d’ingestion déploie les trois notebooks répertoriés dans Notebooks d’ingestion des données DICOM. Certains paramètres de configuration du notebook sont hérités de la configuration globale et peuvent être remplacés au niveau du notebook. Les notebooks incluent également des paramètres de configuration spécifiques définis dans le dictionnaire kwargs. Vous pouvez fournir ces paramètres lorsque vous appelez la bibliothèque de services dans le code du notebook.
Configurer l’ingestion des données à partir des fichiers FHIR ImagingStudy
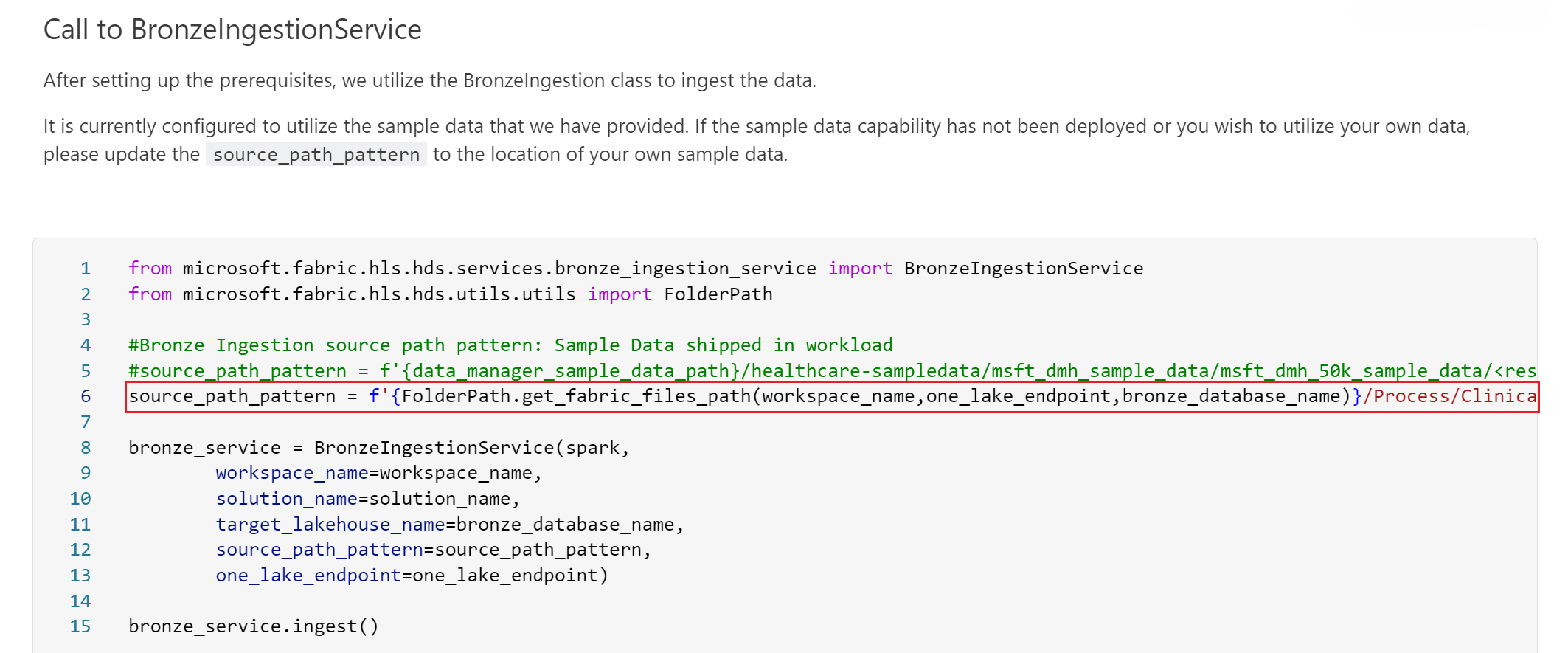
Pour configurer l’ingestion des données à partir des fichiers FHIR ImagingStudy, vous devez modifier la valeur du paramètre source_path_pattern dans le notebook healthcare#_msft_raw_bronze_ingestion. Cette modification est nécessaire puisque la valeur préconfigurée de ce paramètre fait référence au dossier des exemples de données cliniques.
Reconfigurez la valeur de ce paramètre pour faire référence à la structure de dossiers optimisée pour vos données d’imagerie médicale sous Files\Process\Clinical\FHIR NDJSON\Fabric.HDS\yyyy\mm\dd\ImagingStudy. Par exemple, votre paramètre source_path_pattern doit ressembler à la valeur suivante :
source_path_pattern = f'{FolderPath.get_fabric_files_path(workspace_name,one_lake_endpoint,bronze_database_name)}/Process/Clinical/FHIR NDJSON/Fabric.HDS/**/**/**/ImagingStudy/<resource_name>*ndjson'
Configurer l’ingestion Azure Data Lake Storage
Suivez cette option de configuration Bring Your Own Storage (BYOS) si vous souhaitez utiliser les fichiers DICOM à partir de votre emplacement de stockage Azure Data Lake Storage Gen2. Avec cette option, vous n’avez pas besoin de copier ou de déplacer les fichiers DICOM vers les dossiers d’ingestion OneLake. À la place, vous pouvez créer un raccourci OneLake vers Data Lake Storage Gen2 et accéder aux fichiers DICOM à partir de leur emplacement de stockage d’origine. Pour consulter les étapes détaillées et le pipeline d’exécution, accédez à Option 2 : Ingestion des données DICOM à partir de Azure Data Lake Storage Gen2.
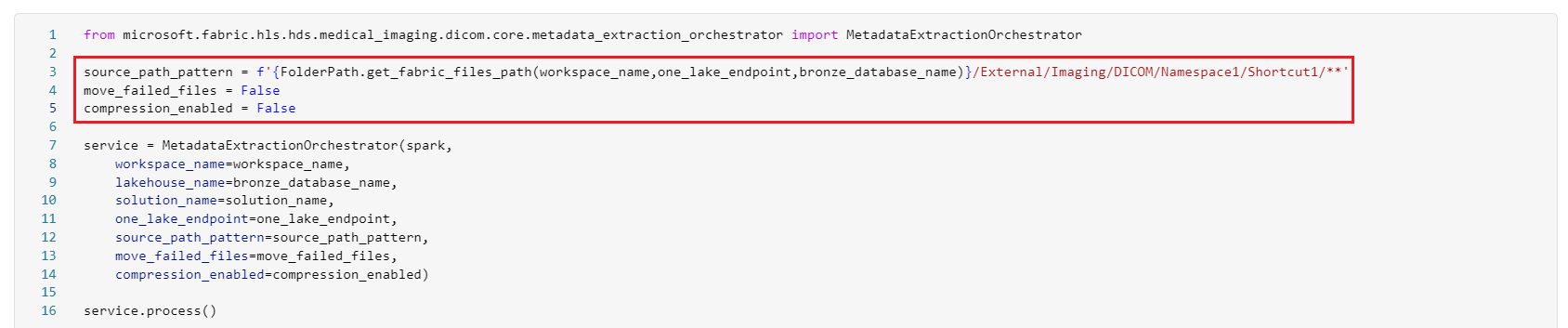
Pour configurer cette option d’ingestion, reconfigurez les paramètres du notebook healthcare#_msft_imaging_dicom_extract_bronze_ingestion. Cette reconfiguration désactive la compression ou le déplacement de fichiers vers les dossiers en échec et redirige le notebook vers le chemin de raccourci sous Files\External\Imaging\DICOM\[Namespace]\[ShortcutName].
Reconfigurez les paramètres du notebook comme suit :
source_path_pattern = f'{FolderPath.get_fabric_files_path(workspace_name,one_lake_endpoint,bronze_database_name)}/External/Imaging/DICOM/Namespace1/Shortcut1/**move_failed_files = Falsecompression_enabled = False
Configurer l’ingestion des données à partir des fichiers FHIR DiagnosticReport
Important
Il existe un problème connu avec les rapports de diagnostic dans les exemples de données. Les rapports de diagnostic sont actuellement déployés au format JSON au lieu du format NDJSON. Vous ne pouvez pas ingérer ces fichiers tels quels ; ils doivent d’abord être convertis au format NDJSON. Nous travaillerons à la résolution de ce problème dans la prochaine version.
Pour ingérer les fichiers NDJSON DiagnosticReport FHIR dans la lakehouse bronze, modifiez le paramètre source_path_pattern dans le notebook healthcare#_msft_raw_bronze_ingestion. Mettez à jour la valeur du paramètre pour qu’elle pointe vers la structure de dossiers optimisée pour les rapports de diagnostic sous Files\Process\Clinical\FHIR NDJSON\Fabric.HDS\yyyy\mm\dd\DiagnosticReport.
Par exemple, votre paramètre source_path_pattern doit ressembler à la valeur suivante :
source_path_pattern = f'{FolderPath.get_fabric_files_path(workspace_name,one_lake_endpoint,bronze_database_name)}/Process/Clinical/FHIR NDJSON/Fabric.HDS/**/**/**/DiagnosticReport/<resource_name>*ndjson
Voir aussi
- Vue d’ensemble des solutions de données de santé dans Microsoft Fabric (version préliminaire)
- Déployer les solutions de données de santé dans Microsoft Fabric (version préliminaire)
- Vue d’ensemble de l’ingestion des données DICOM
- Utiliser l’ingestion des données DICOM
- Transformation des métadonnées DICOM dans les solutions de données de santé (version préliminaire)